Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Ziele für Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Produkte für Chromatin/Epigenetics
- Bestell-Nr. Artikelname Informationen
-
GC65326
GNE-375
GNE-375 ist ein potenter und hochselektiver BRD9-Inhibitor mit einem IC50 von 5 nM. GNE-375 zeigt >100-fach selektiv fÜr BRD9 gegenÜber BRD4, TAF1 und CECR2. GNE-375 verringert die Bindung von BRD9 an Chromatin.
-
GC32081
GNE-781
GNE-781 ist ein oral aktiver, hochpotenter und selektiver CBP-Inhibitor mit einem IC50-Wert von 0,94 nM im TR-FRET-Assay. GNE-781 hemmt auch BRET und BRD4(1) mit IC50-Werten von 6,2 nM bzw. 5100 nM. GNE-781 zeigt AntitumoraktivitÄt in einem MOLM-16-AML-Xenotransplantatmodell.
-
GC33253
GNE-955
GNE-955 ist ein potenter und oral aktiver Pan-Pim-Kinase-Inhibitor mit Kis von 0,018, 0,11, 0,08 nM fÜr Pim1, Pim2 bzw. Pim3.
-
GC40918
Gramicidin A
Gramicidin A ist ein Peptidbestandteil von Gramicidin, einem ursprÜnglich aus B.
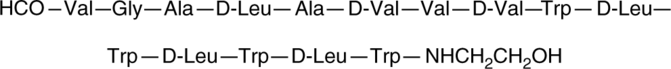
-
GC46152
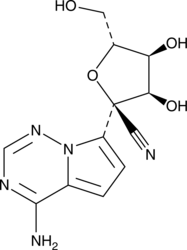
GS-441524
GS-441524, der vorherrschende Metabolit von Remdesivir und Remdesivir gegenÜber Covid-19 Überlegen, zeigt eine vergleichbare Wirksamkeit in zellbasierten Modellen primÄrer menschlicher Lungen- und Katzenzellen, die mit Coronavirus infiziert sind.
-
GC33204
GS-626510
GS-626510 ist ein potenter und oral aktiver BromodomÄnen-Inhibitor der BET-Familie mit Kd-Werten von 0,59-3,2 nM fÜr BRD2/3/4, mit IC50-Werten von 83 nM und 78 nM fÜr BD1 bzw. BD2.
-
GC31731
GSK 4027
GSK 4027 ist eine chemische Sonde fÜr die PCAF/GCN5-BromodomÄne mit einem pIC50 von 7,4 ± 0,11 fÜr PCAF in einem zeitaufgelÖsten Fluoreszenz-Resonanz-Energietransfer (TR-FRET)-Assay.
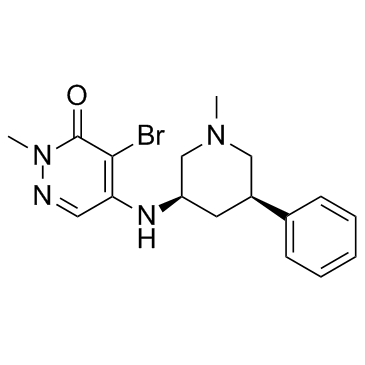
-
GC10524
GSK 525768A
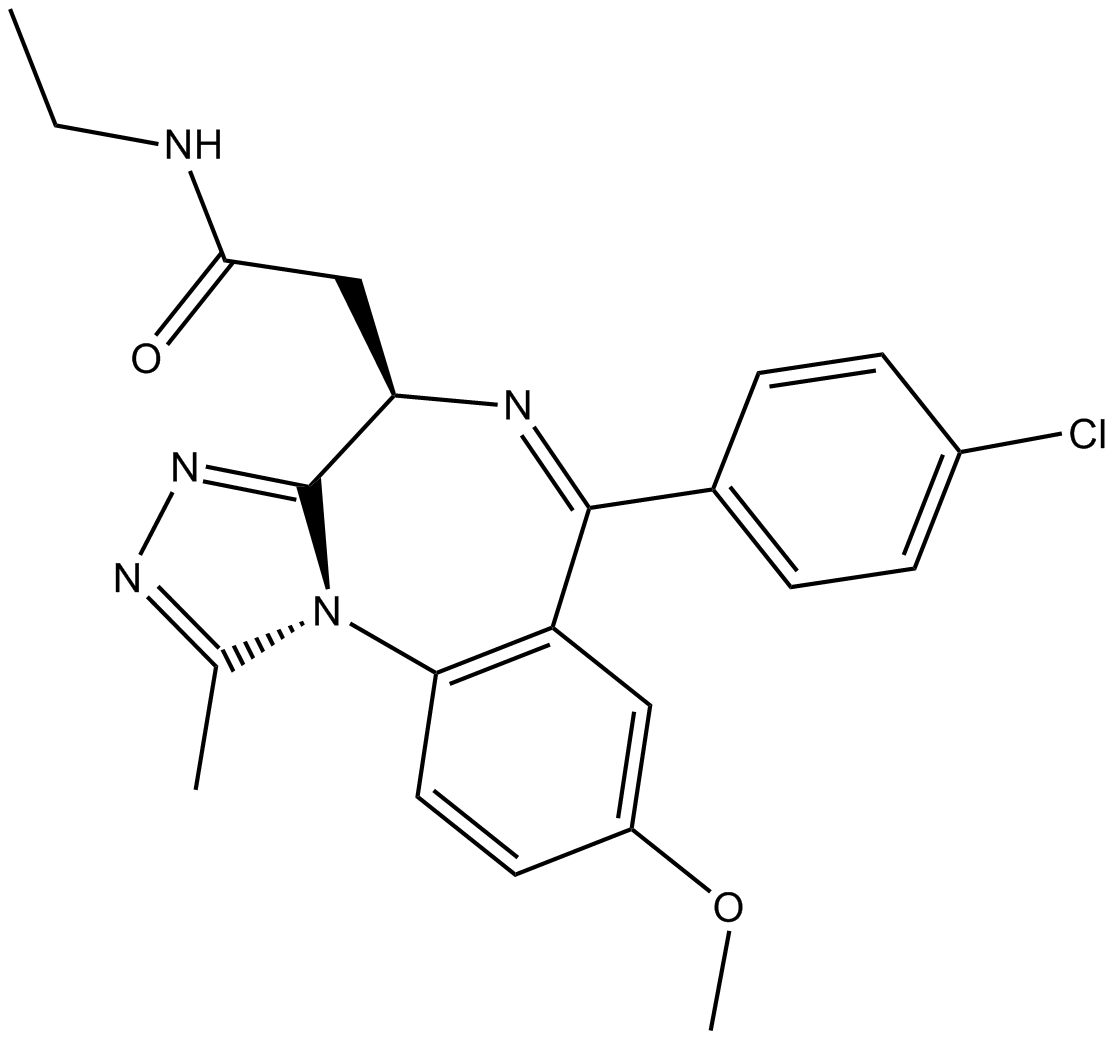
-
GC50697
GSK 591 dihydrochloride
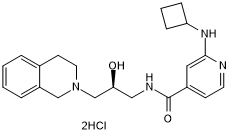
-
GC12440
GSK 5959
GSK 5959 ist ein potenter, selektiver und zellgängiger BRPF1-Bromodomänen-Inhibitor mit einem IC50 von ~ 80 nM.
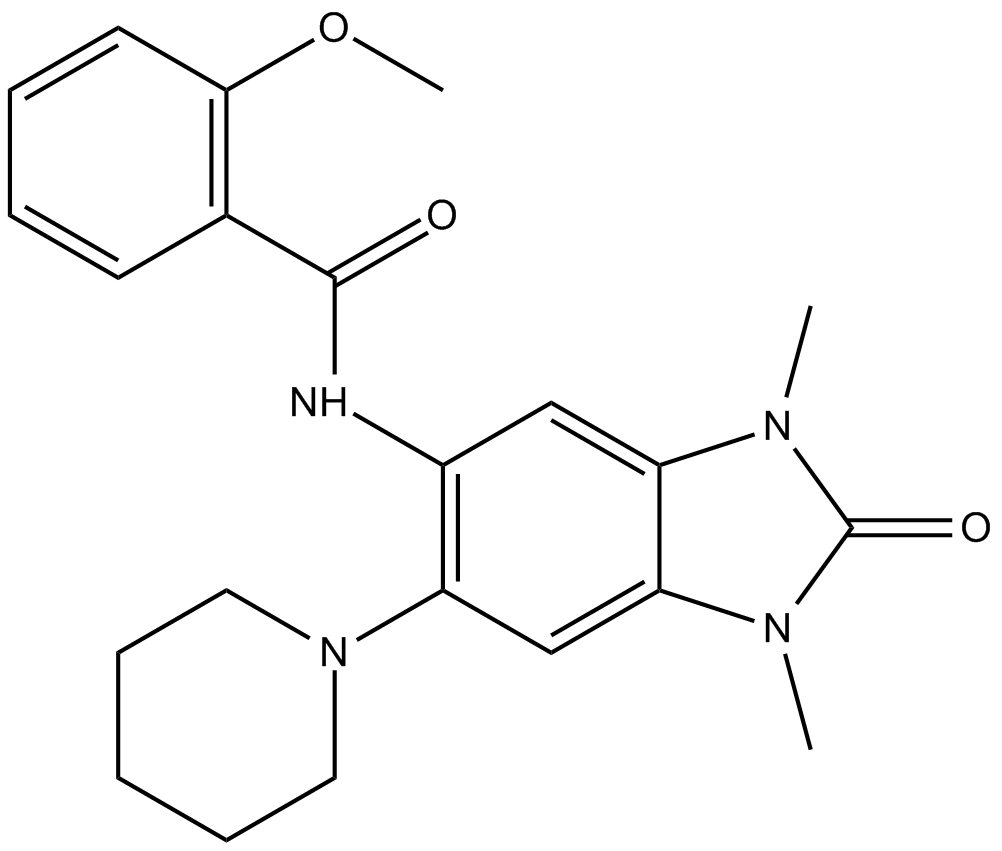
-
GC34314
GSK 690 Hydrochloride
GSK 690 (Hydrochlorid) ist ein reversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1) mit einem Kd-Wert von 9 nM und einem biochemischen IC50-Wert von 37 nM.
-
GC50378
GSK 9311 hydrochloride
GSK 9311-Hydrochlorid, ein weniger aktives Analogon von GSK6853, kann als Negativkontrolle verwendet werden. GSK 9311-Hydrochlorid hemmt die BRPF-Bromodomäne mit pIC50-Werten von 6,0 und 4,3 für BRPF1 bzw. BRPF2.
-
GC10617
GSK J1
A dual inhibitor of JMJD3 and UTX
-
GC13086
GSK J2
GSK J2 ist ein Isomer von GSK-J1, das keine spezifische Aktivität aufweist. GSK-J1 ist ein potenter Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A.
-
GC12997
GSK J4 free base
Die freie Base GSK J4 ist ein potenter dualer Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A mit IC50-Werten von 8,6 bzw. 6,6 μM. Die freie Base von GSK J4 inhibiert LPS-induziertes TNF-⋱ Produktion in humanen primÄren Makrophagen mit einem IC50 von 9 μM. GSK J4 ist ein zelldurchlÄssiges Prodrug von GSK-J1. Die freie Base von GSK J4 induziert eine stressbedingte Apoptose des endoplasmatischen Retikulums.
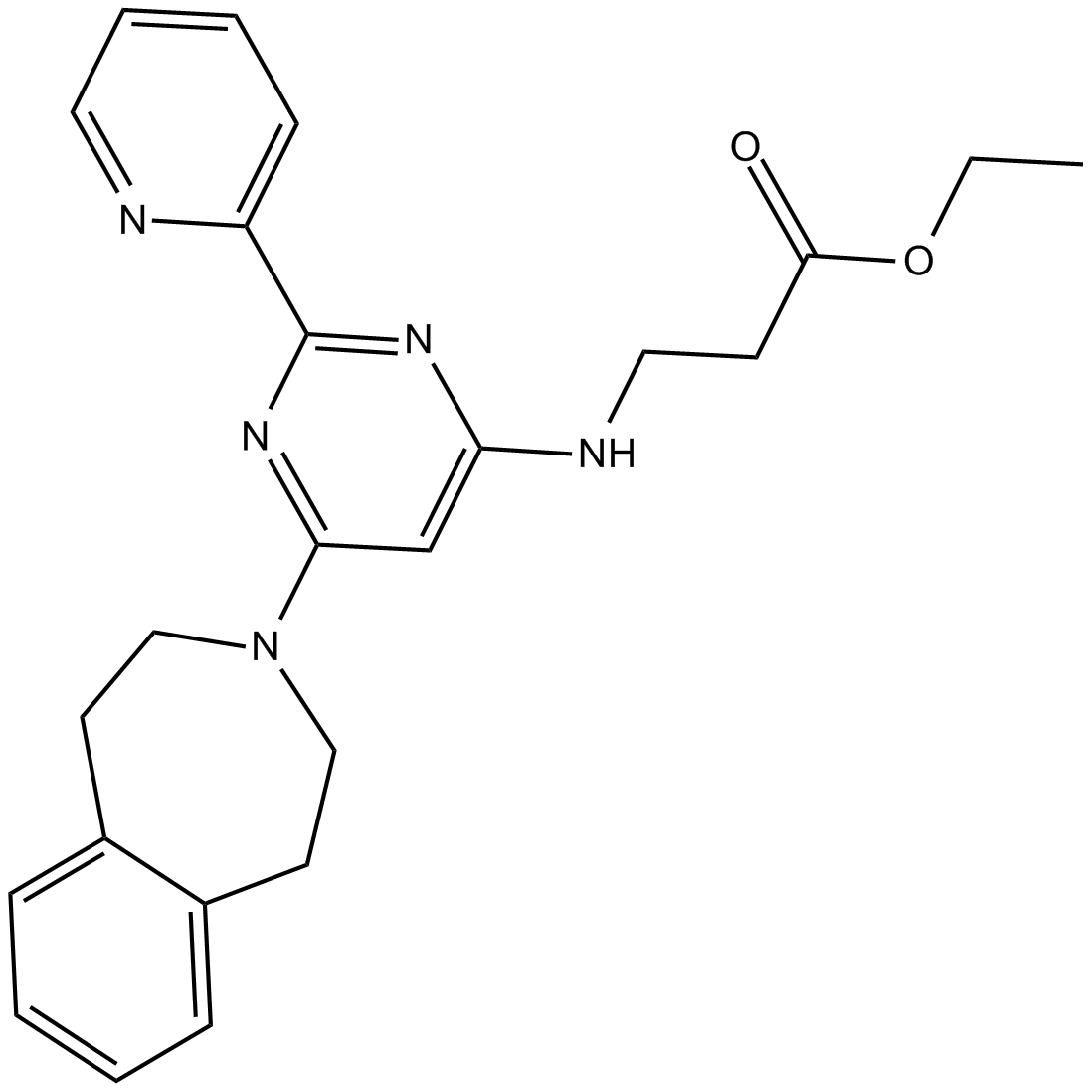
-
GC15497
GSK J4 HCl
Prodrug eines selektiven H3K27-Histon-Demethylase-Inhibitors.
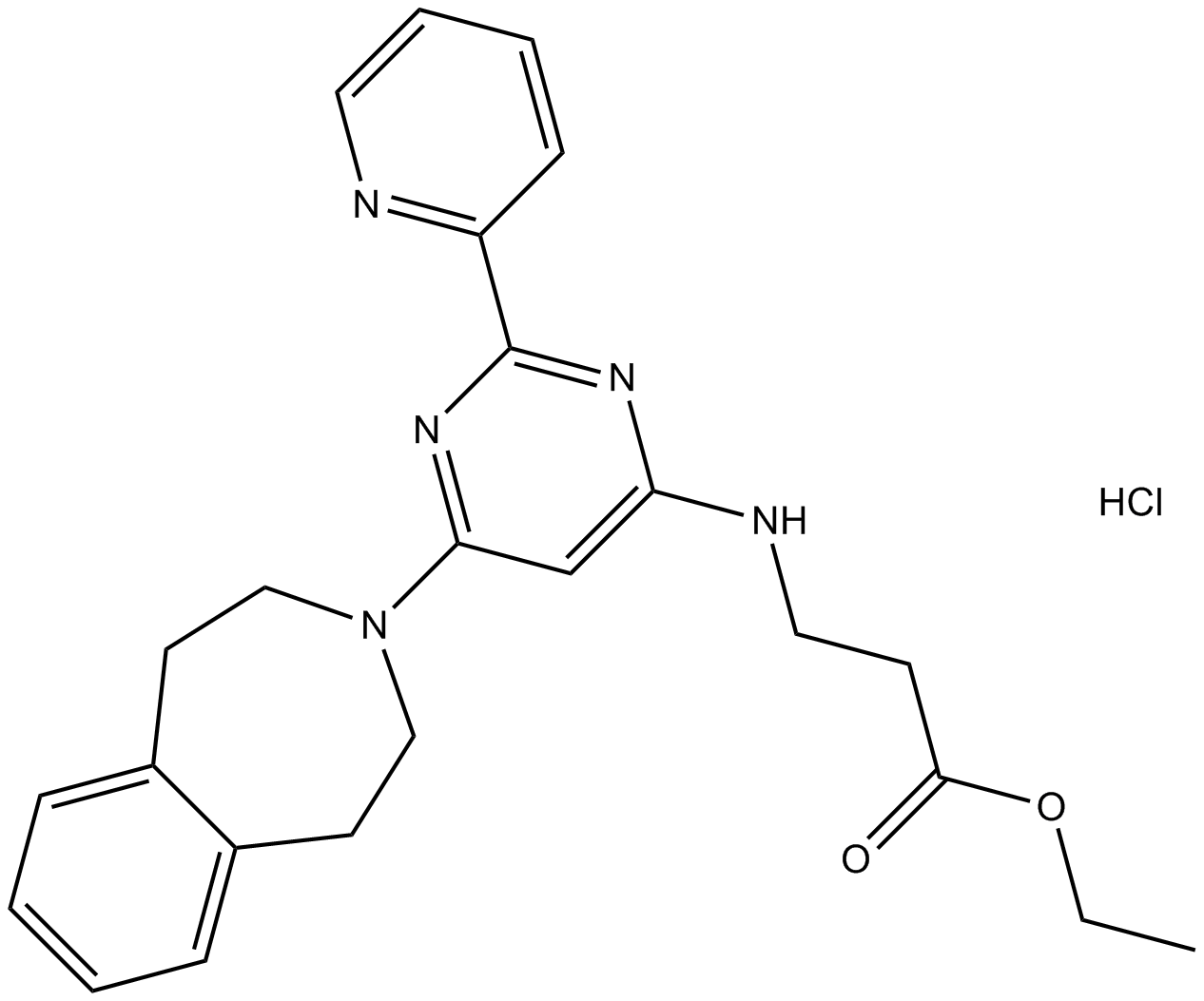
-
GC17118
GSK J5
inactive isomer of GSK J4, KDM inhibitor
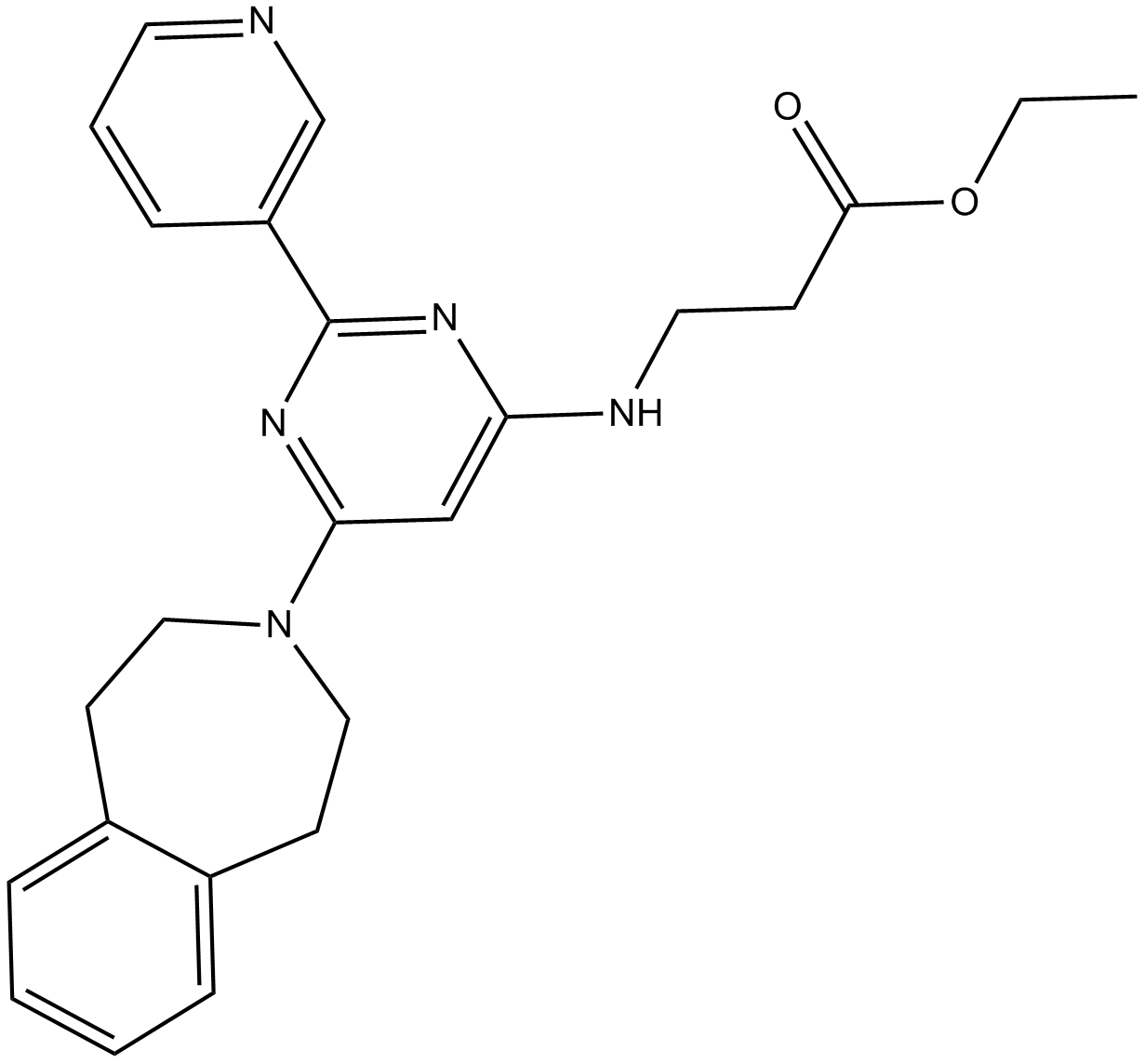
-
GC63483
GSK-3685032
GSK-3685032 ist ein nicht zeitabhÄngiger, nicht kovalenter, erster reversibler DNMT1-selektiver Inhibitor seiner Klasse mit einem IC50-Wert von 0,036 μM. GSK-3685032 induziert einen starken Verlust der DNA-Methylierung, Transkriptionsaktivierung und Wachstumshemmung von Krebszellen.
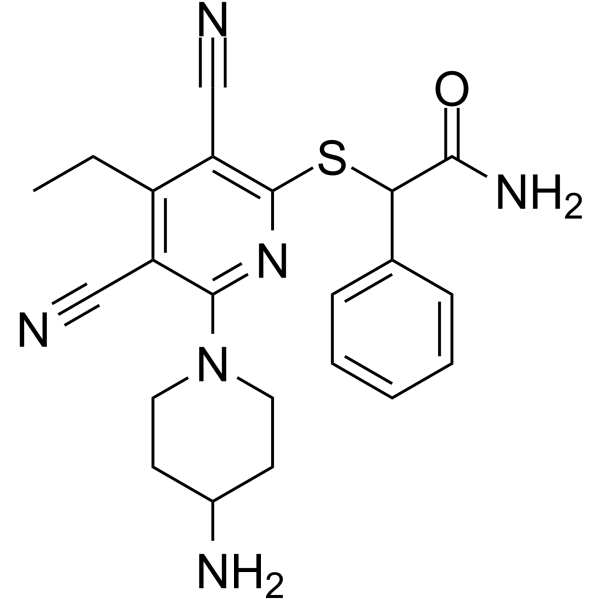
-
GC36195
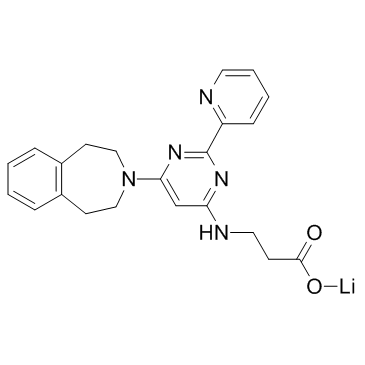
GSK-J1 lithium salt
GSK-J1-Lithiumsalz ist ein potenter Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A mit einem IC50-Wert von 60 nM gegenÜber KDM6B.
-
GC43792
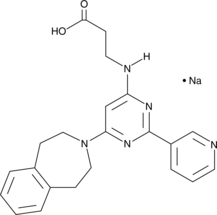
GSK-J2 (sodium salt)
The histone H3 lysine 27 (H3K27) demethylase JMJD3 plays important roles in the transcriptional regulation of cell differentiation, development, the inflammatory response, and cancer.
-
GC43794
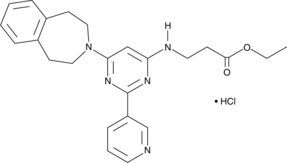
GSK-J5 (hydrochloride)
GSK-J5 (Hydrochlorid) ist ein zellgängiges Esterderivat von GSK J2, inaktiv. GSK-J5 (Hydrochlorid) ist ebenfalls ein Isomer von GSK-J4 und wird häufig als negative Gruppe verwendet.
-
GC18012
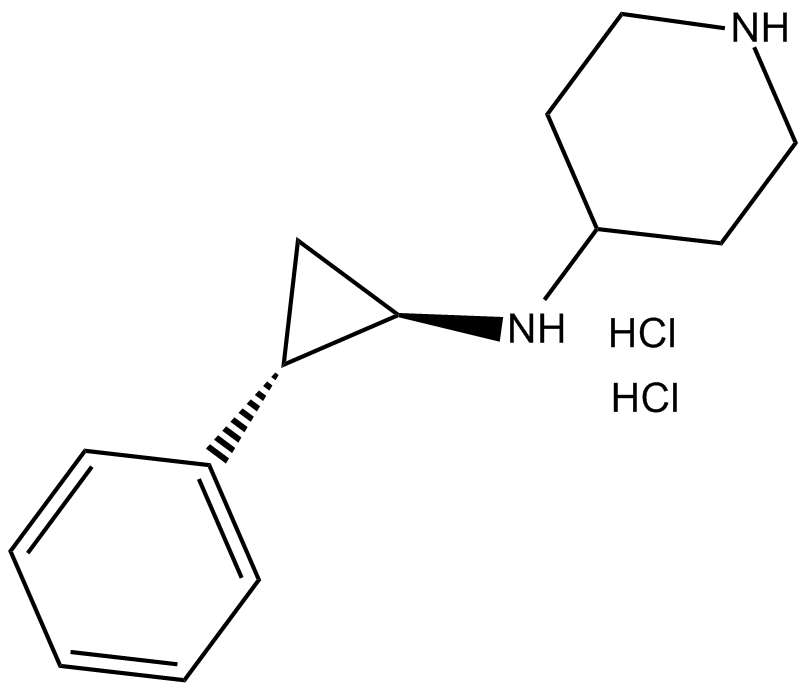
GSK-LSD1 (hydrochloride)
LSD1 inhibitor
-
GC15368
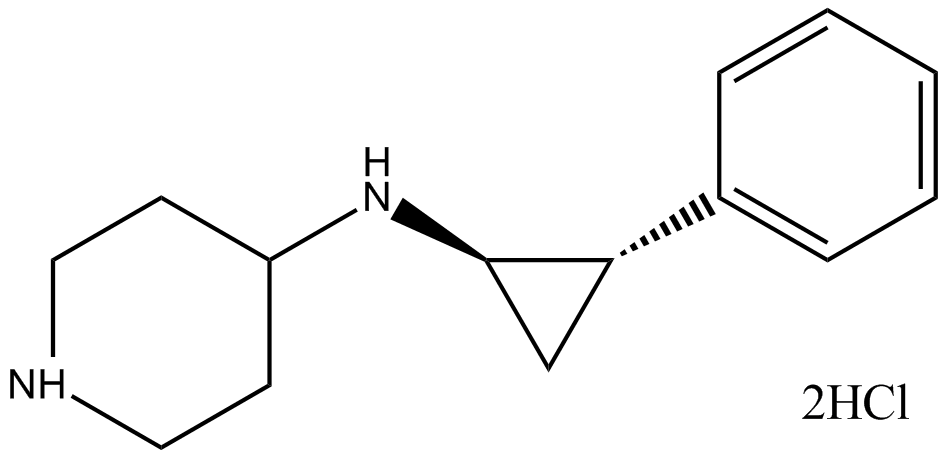
GSK-LSD1 2HCl
irreversible, and selective LSD1 inhibitor
-
GC32764
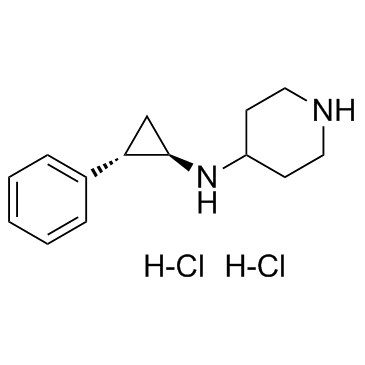
GSK-LSD1 Dihydrochloride
GSK-LSD1-Dihydrochlorid ist ein potenter, selektiver und irreversibler Hemmer der Lysin-spezifischen Demethylase 1 (LSD1) mit einem IC50-Wert von 16 nM.
-
GC43787
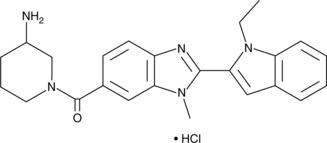
GSK106 (hydrochloride)
GSK106 (Hydrochlorid) ist eine inaktive Kontrolle fÜr die selektiven PAD4-Inhibitoren GSK484 und GSK199.
-
GC10008
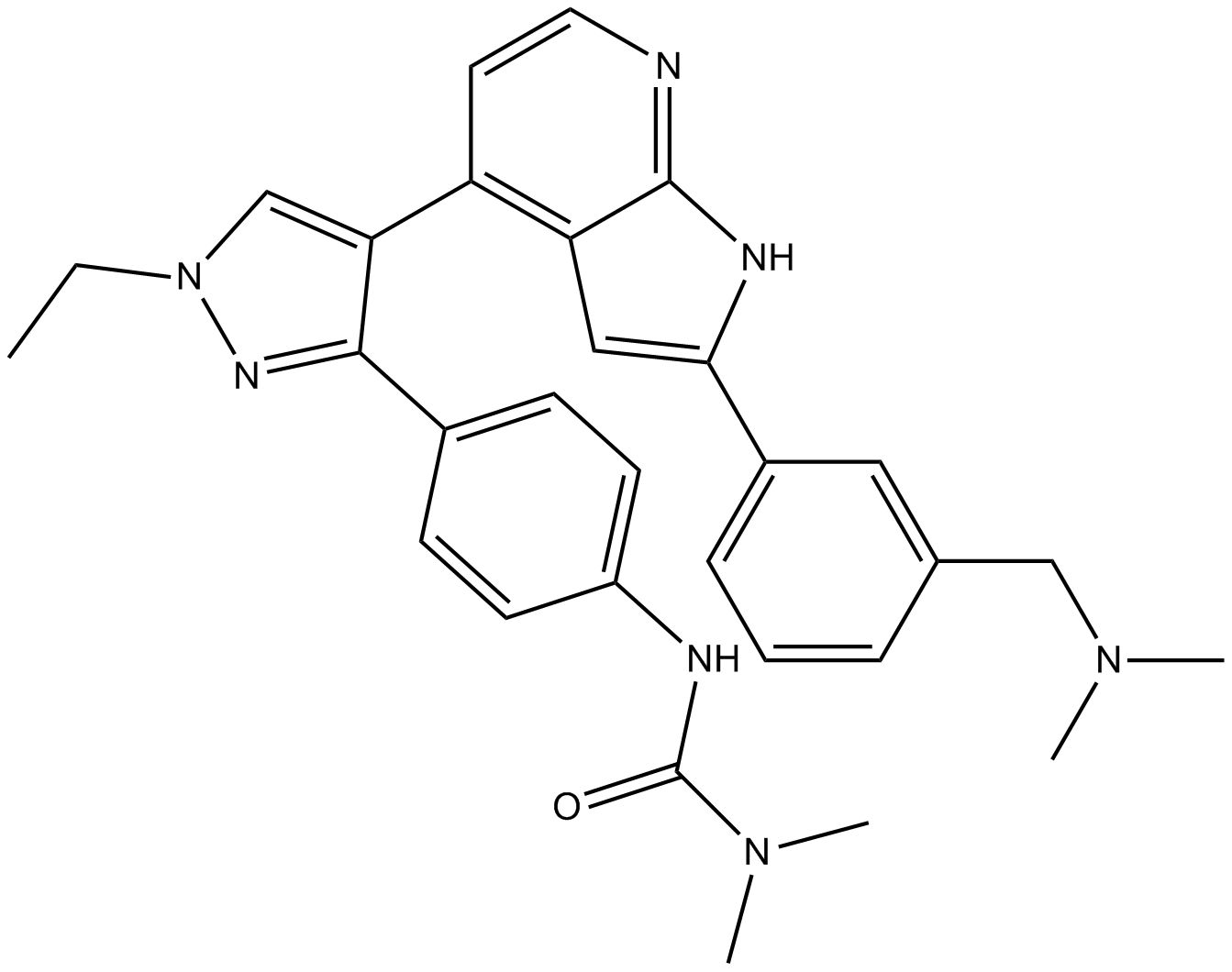
GSK1070916
A potent inhibitor of Aurora B and C kinases
-
GC43788
GSK121 (trifluoroacetate salt)
GSK-121 Trifluoracetate ein selektiver PAD4-Hemmer.
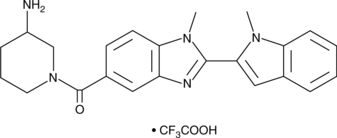
-
GC15783
GSK126
Ein selektiver EZH2-Inhibitor
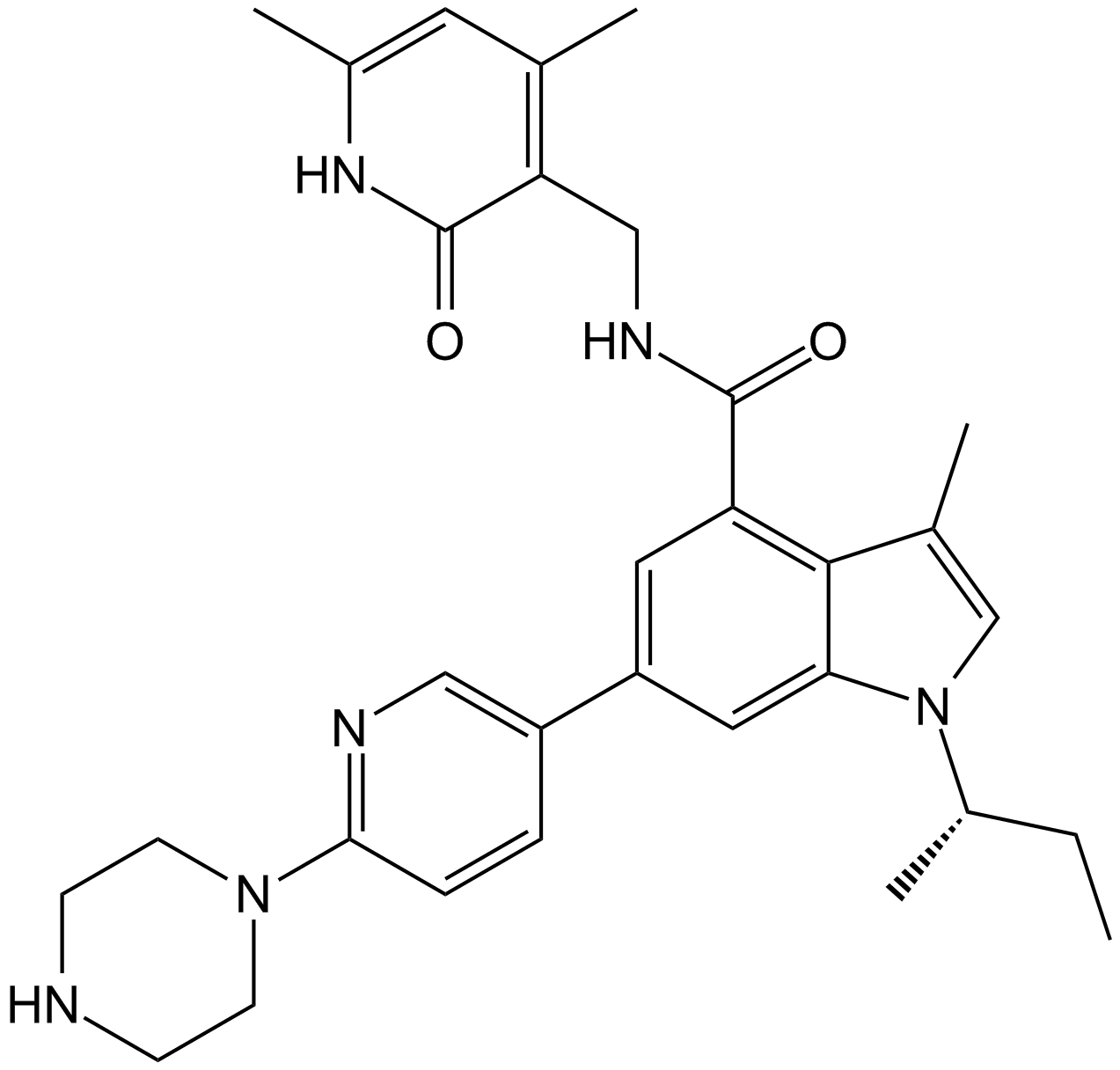
-
GC14063
GSK1324726A
GSK1324726A ist ein neuer, potenter und selektiver Inhibitor von BET-Proteinen mit hoher AffinitÄt zu BRD2 (IC50=41 nM), BRD3 (IC50=31 nM) und BRD4 (IC50=22 nM).
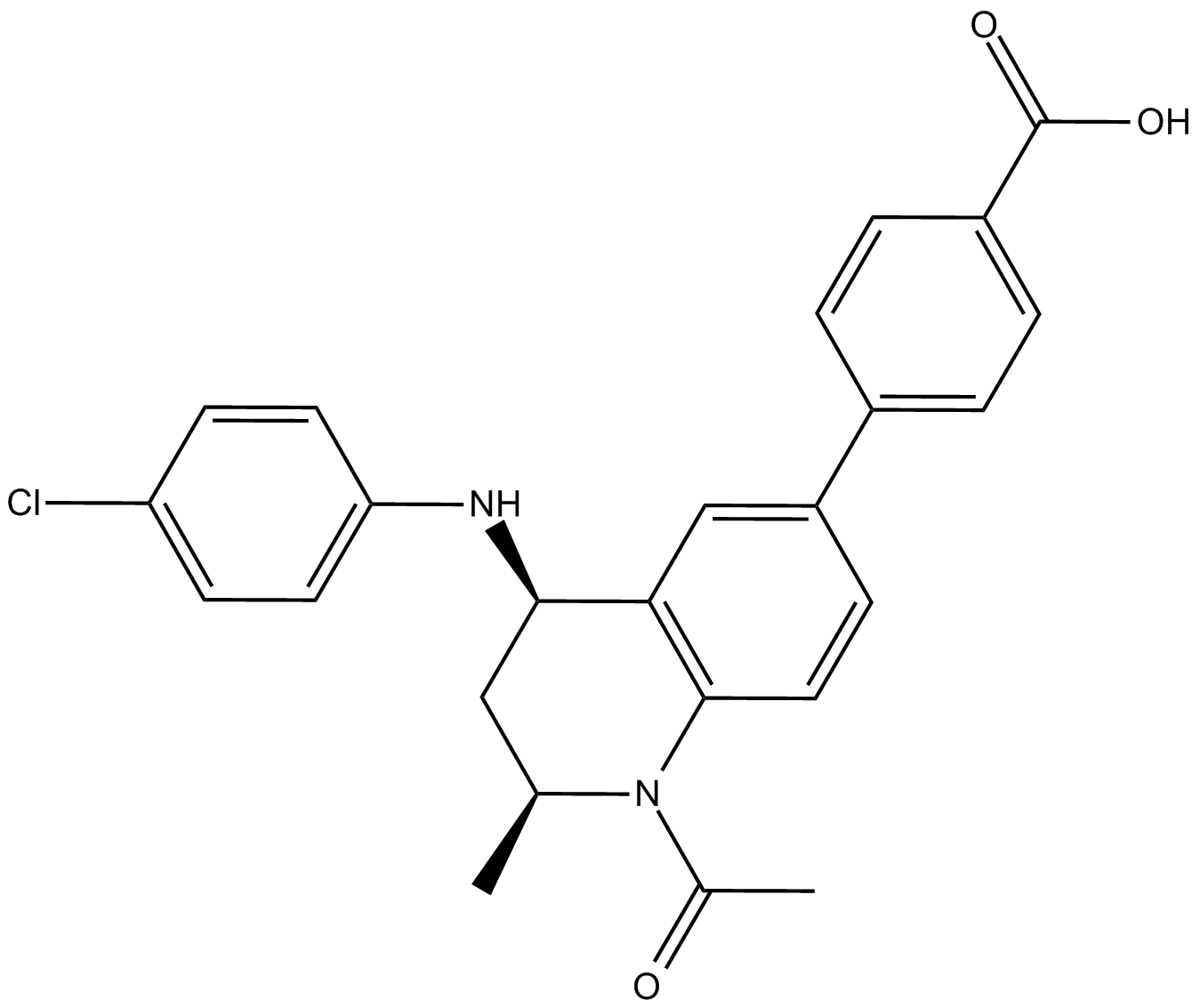
-
GC43789
GSK199 (hydrochloride)
GSK199 (Hydrochlorid) ist ein reversibler und selektiver PAD4-Inhibitor mit einer IC50 von 200 nM in Abwesenheit von Calcium.
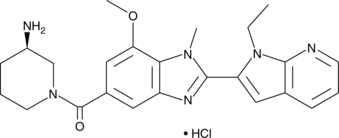
-
GC15789
GSK2801
GSK2801 ist ein potenter, selektiver, oral aktiver und zellaktiver Acetyl-Lysin-kompetitiver BAZ2A- und BAZ2B-BromodomÄnen-Inhibitor mit Kd-Werten von 136 nM bzw. 257 nM. GSK2801 zeigt eine >50-fache SelektivitÄt fÜr BAZ2A/B gegenÜber BRD4.
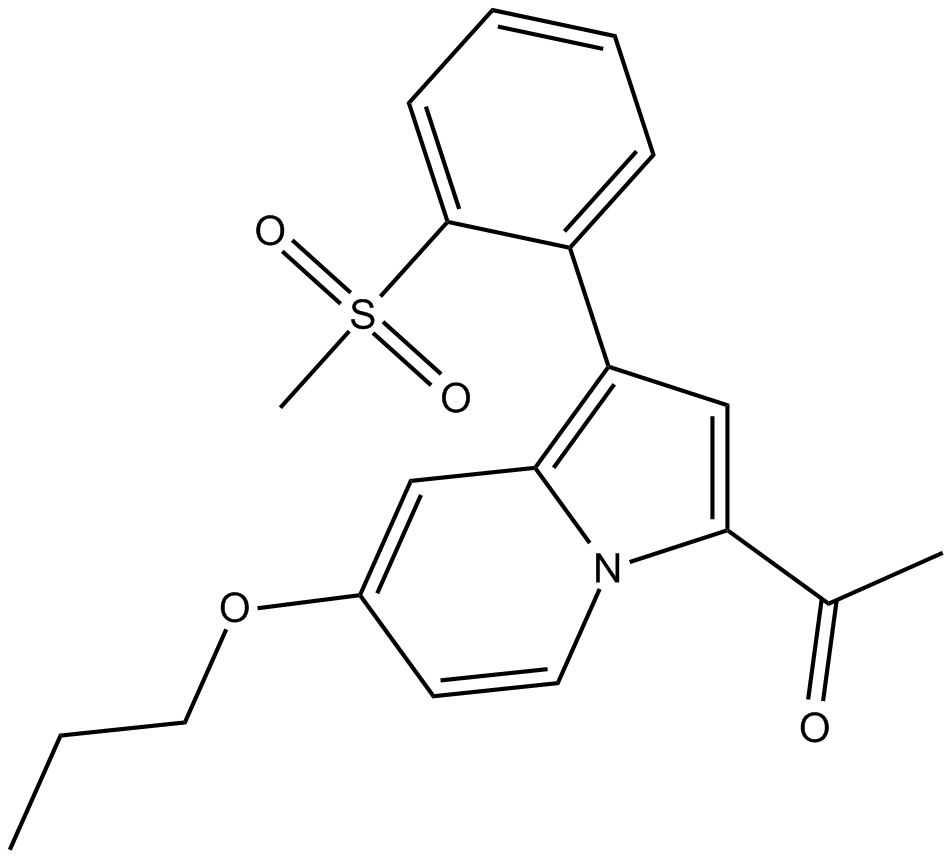
-
GC19181
GSK2807 Trifluoroacetate
GSK2807 Trifluoracetat ist ein potenter, selektiver und SAM-kompetitiver Inhibitor von SMYD3 mit einem Ki von 14 nM und einem IC50 von 130 nM.
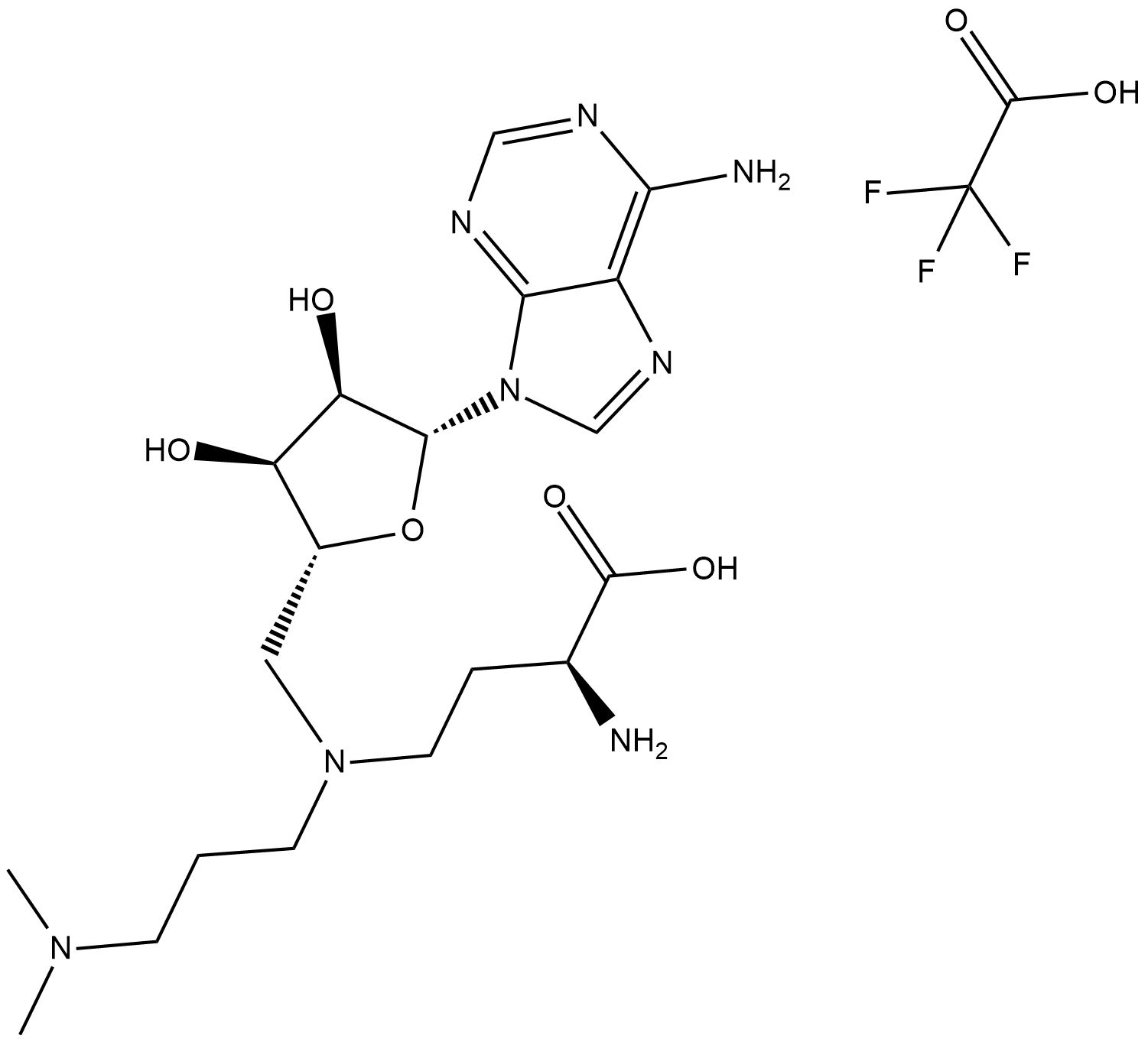
-
GC11578
GSK2879552
GSK2879552 ein oral aktiver, selektiver und irreversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1/KDM1A) mit potenzieller antineoplastischer AktivitÄt.
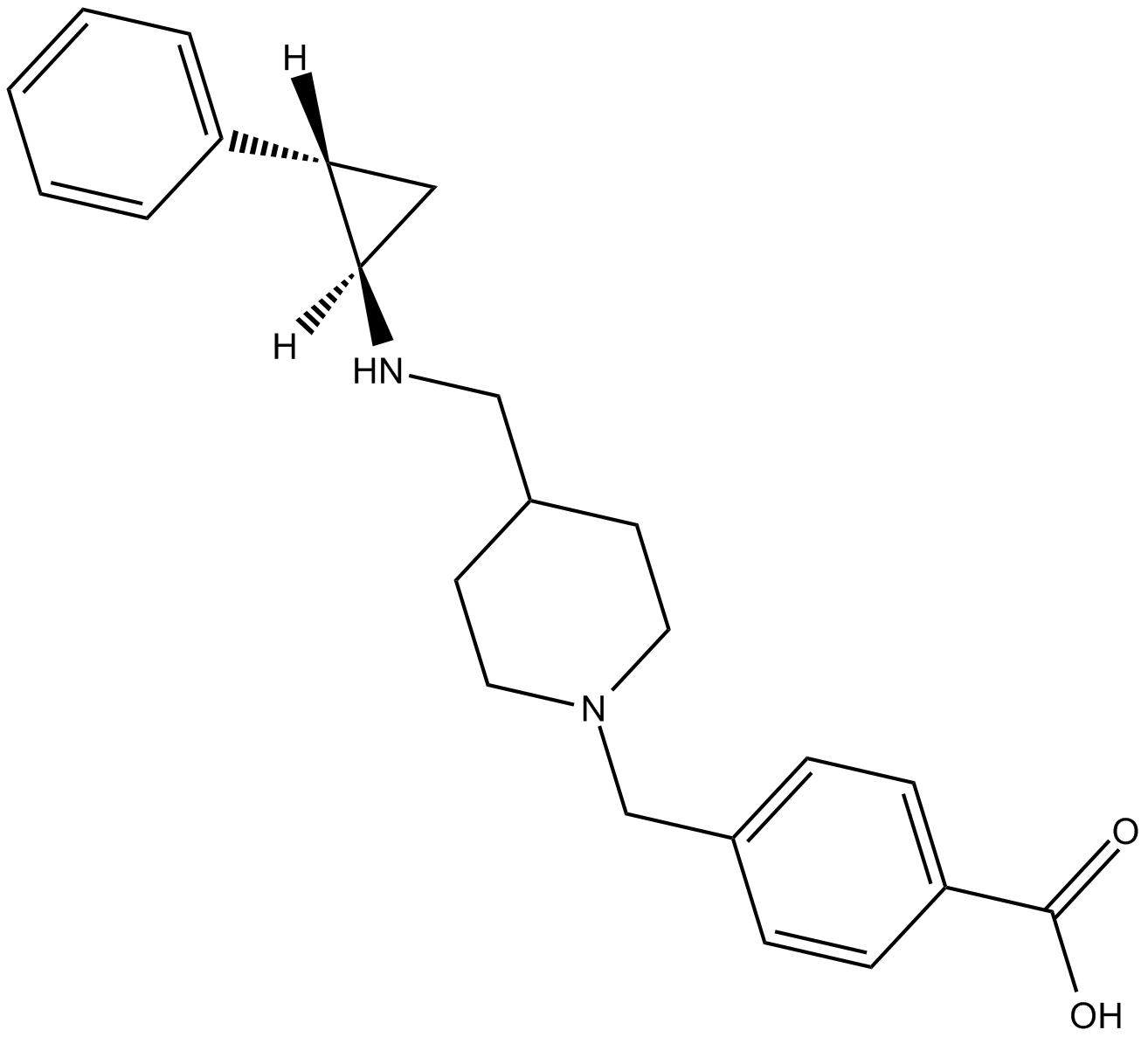
-
GC62719
GSK2879552 dihydrochloride
GSK2879552 Dihydrochlorid, ein oral aktiver, selektiver und irreversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1/KDM1A) mit potenzieller antineoplastischer AktivitÄt.
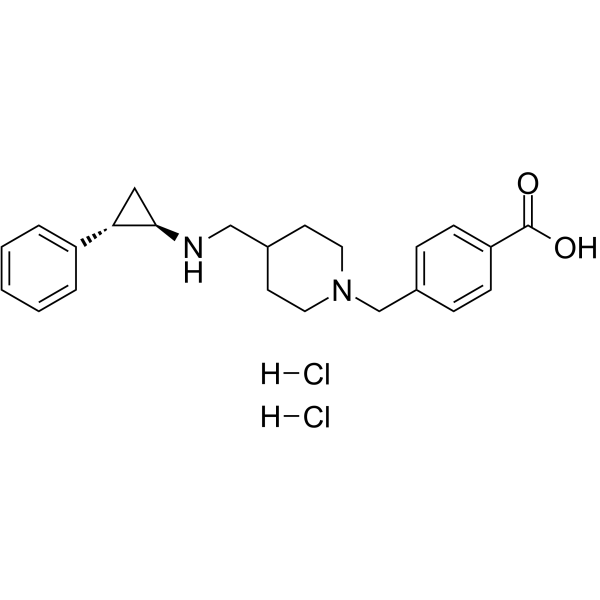
-
GC18402
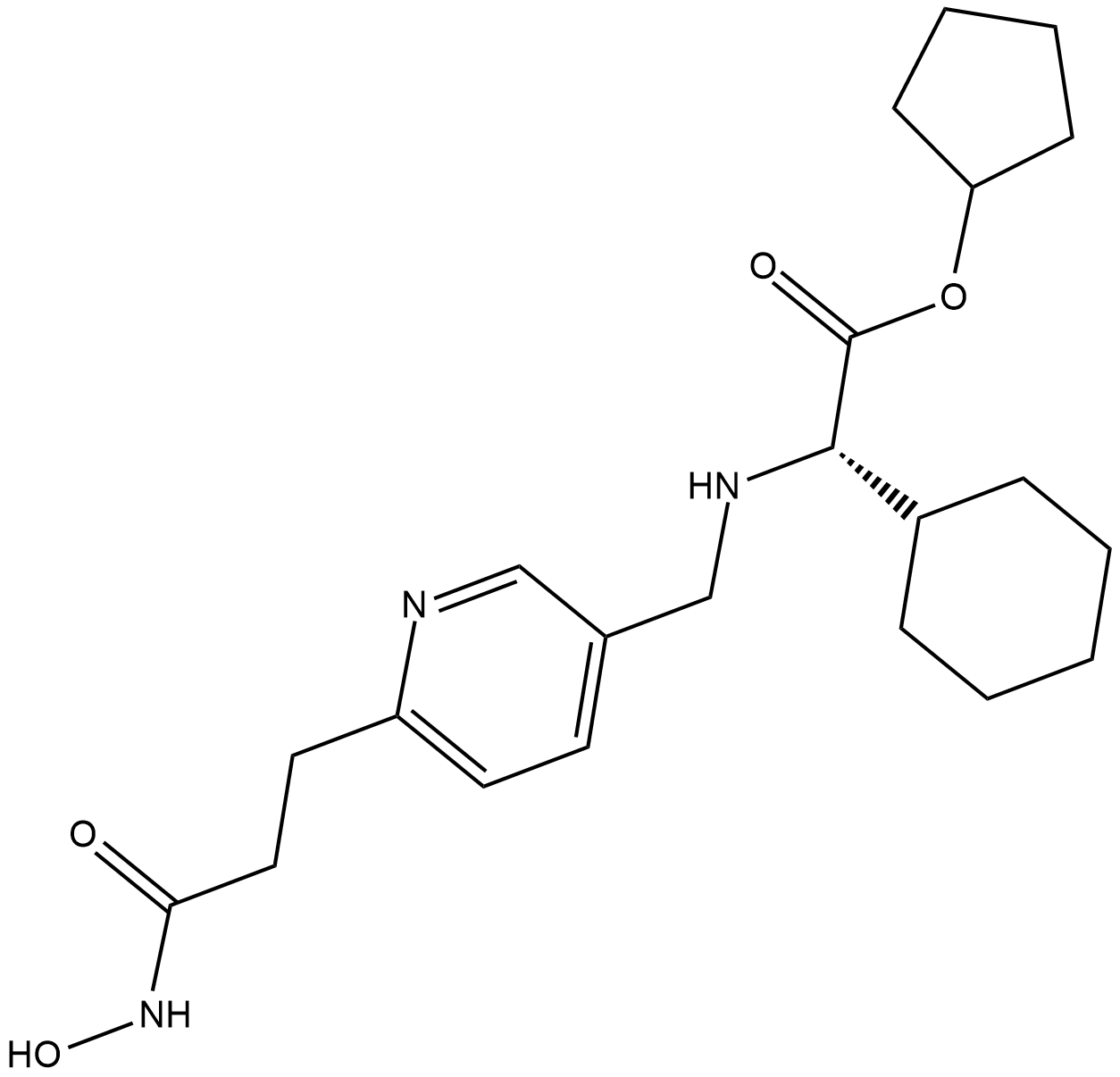
GSK3117391
GSK3117391 ist ein Histon-Deacetylase (HDAC)-Inhibitor, extrahiert aus dem Patent WO/2008040934 A1.
-
GC32693
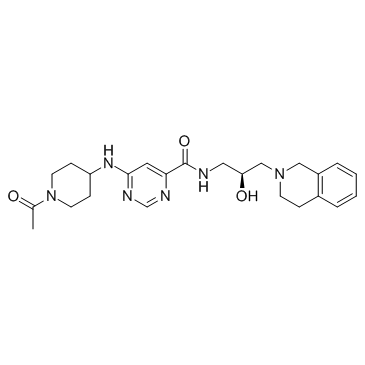
GSK3326595 (EPZ015938)
GSK3326595 (EPZ015938) (EPZ015938) ist ein potenter, selektiver, reversibler Inhibitor der Protein-Arginin-Methyltransferase 5 (PRMT5) mit einem IC50 von 6,2 nM.
-
GC36191
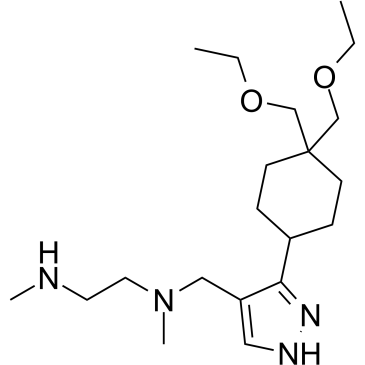
GSK3368715
GSK3368715 (EPZ019997) ist ein oral aktiver, reversibler und S-Adenosyl-L-Methionin (SAM) nicht kompetitiver Typ-I-Protein-Arginin-Methyltransferase (PRMTs)-Inhibitor (IC50=3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM ( PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715 (EPZ019997) bewirkt eine Verschiebung der Arginin-MethylierungszustÄnde, verÄndert die Exon-Nutzung und hat eine starke Anti-Krebs-AktivitÄt.
-
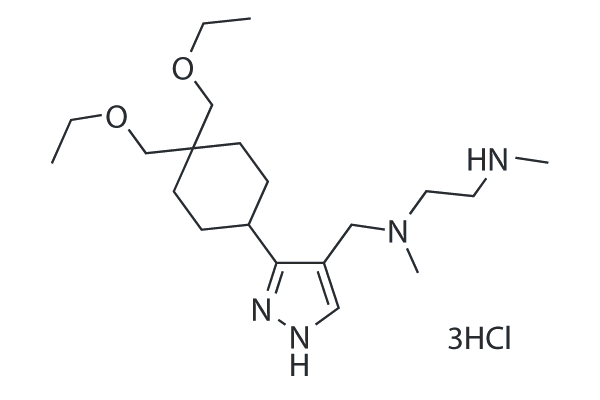
GC25483
GSK3368715 (EPZ019997) 3HCl
GSK3368715 is a potent inhibitor of type I protein arginine methyltransferases (PRMT) that inhibits PRMT1, 3, 4, 6 and 8 with Kiapp vaules ranging from 1.5 to 81 nM.
-
GC49398
GSK3368715 (hydrochloride)
An inhibitor of type I PRMTs
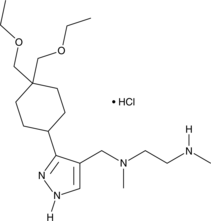
-
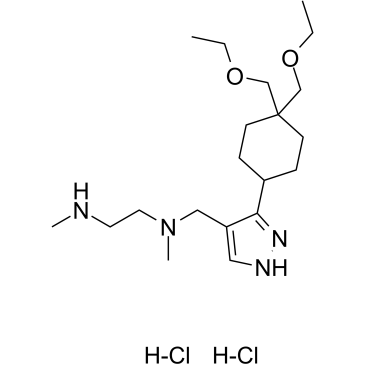
GC36192
GSK3368715 dihydrochloride
GSK3368715 Dihydrochlorid (EPZ019997 Dihydrochlorid) ist ein oral aktiver, reversibler und S-Adenosyl-L-Methionin (SAM) nicht kompetitiver Inhibitor der Typ-I-Protein-Arginin-Methyltransferase (PRMTs) (IC50=3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM (PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715-Dihydrochlorid (EPZ019997-Dihydrochlorid) erzeugt eine Verschiebung der Arginin-MethylierungszustÄnde, verÄndert die Exon-Nutzung und hat eine starke Anti-Krebs-AktivitÄt.
-
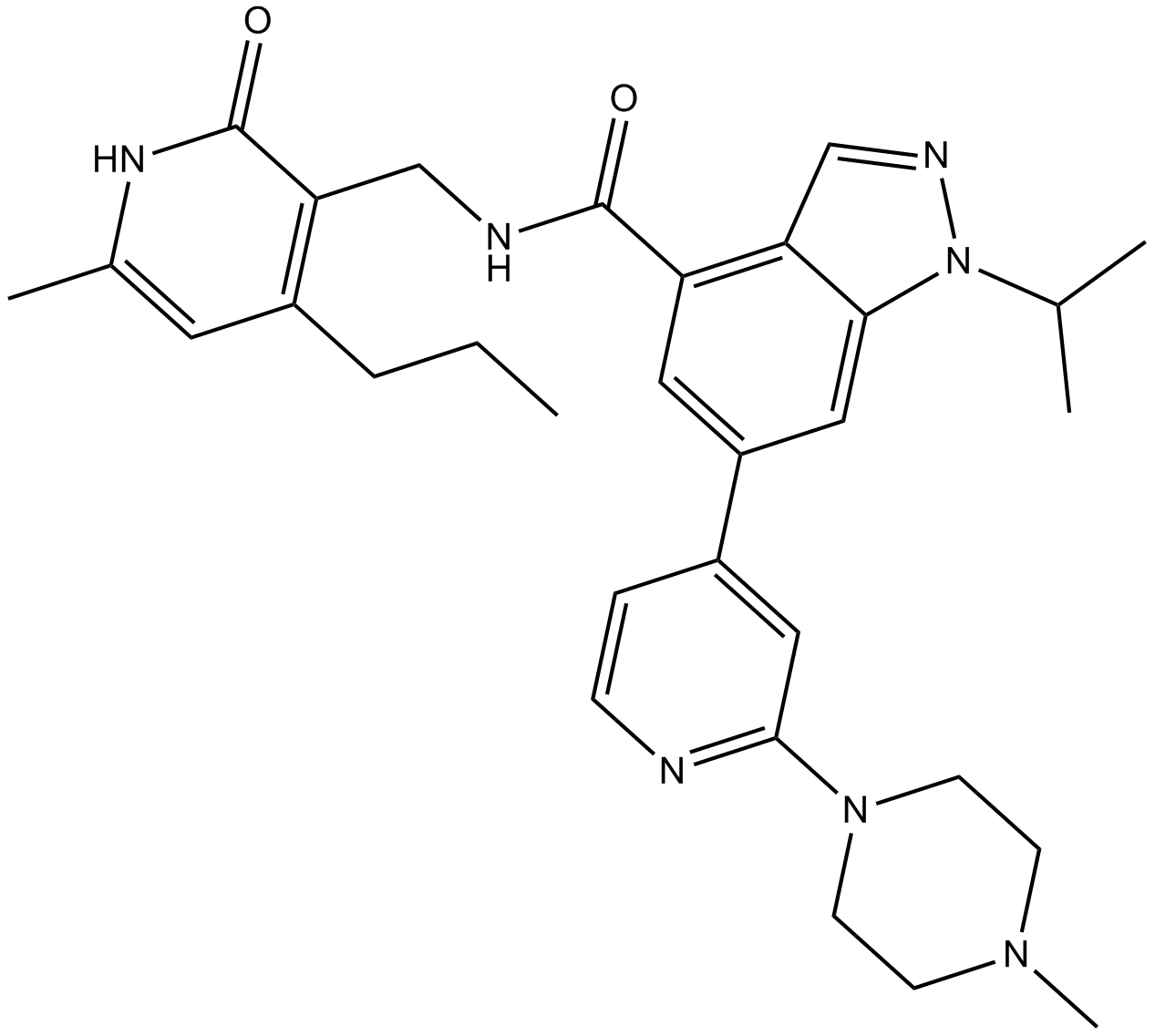
GC17534
GSK343
A selective, cell-permeable EZH2 inhibitor
-
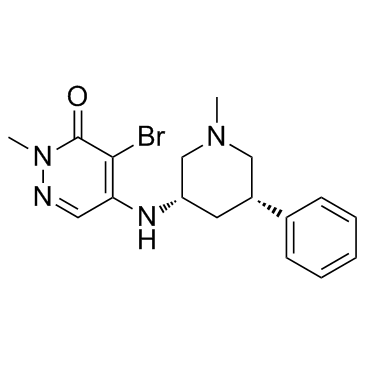
GC30714
GSK4028
GSK4028 ist die enantiomere Negativkontrolle von GSK4027, einer chemischen PCAF/GCN5-BromodomÄnensonde, der pIC50 von GSK4028 betrÄgt 4,9 in einem zeitaufgelÖsten Fluoreszenzresonanzenergietransfer (TR-FRET)-Assay.
-
GC34604
GSK467
GSK467 ist ein zellgÄngiger und selektiver KDM5B (JARID1B oder PLU1)-Inhibitor mit einem Ki von 10 nM und einem IC50 von 26 nM. GSK467 zeigt eine 180-fache SelektivitÄt fÜr KDM4C und keine messbaren inhibitorischen Wirkungen gegenÜber KDM6 oder anderen Mitgliedern der Jumonji-Familie.
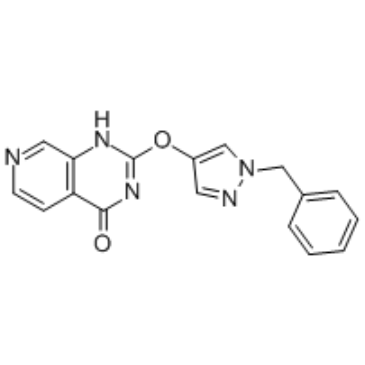
-
GC19184
GSK484 hydrochloride
Ein Peptidylarginin-Deiminase 4 (PAD4)-Inhibitor
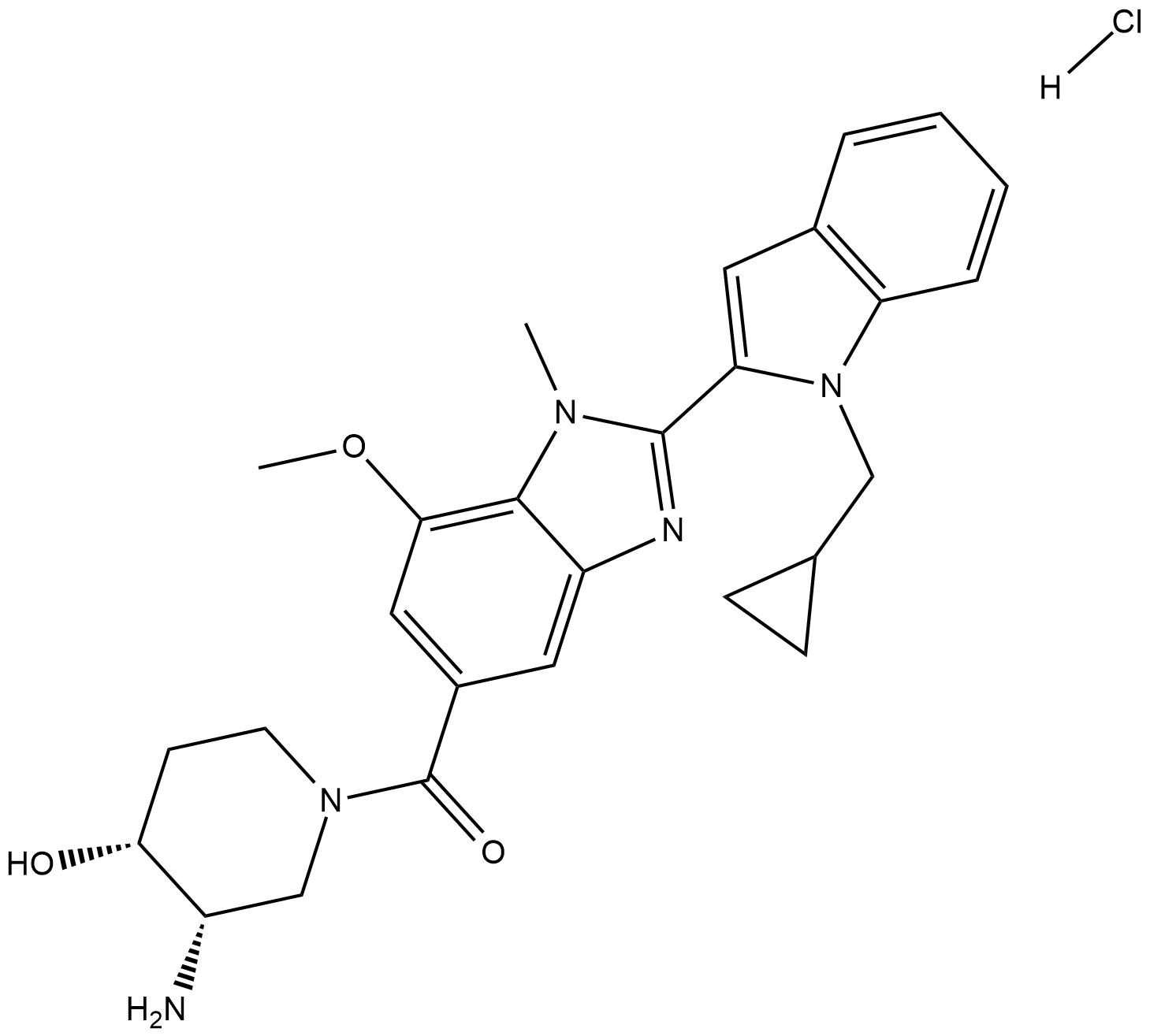
-
GC11414
GSK503
GSK503 ist ein potenter und spezifischer Inhibitor der EZH2-Methyltransferase mit Kiapp-Werten von 3 bis 27 nM.
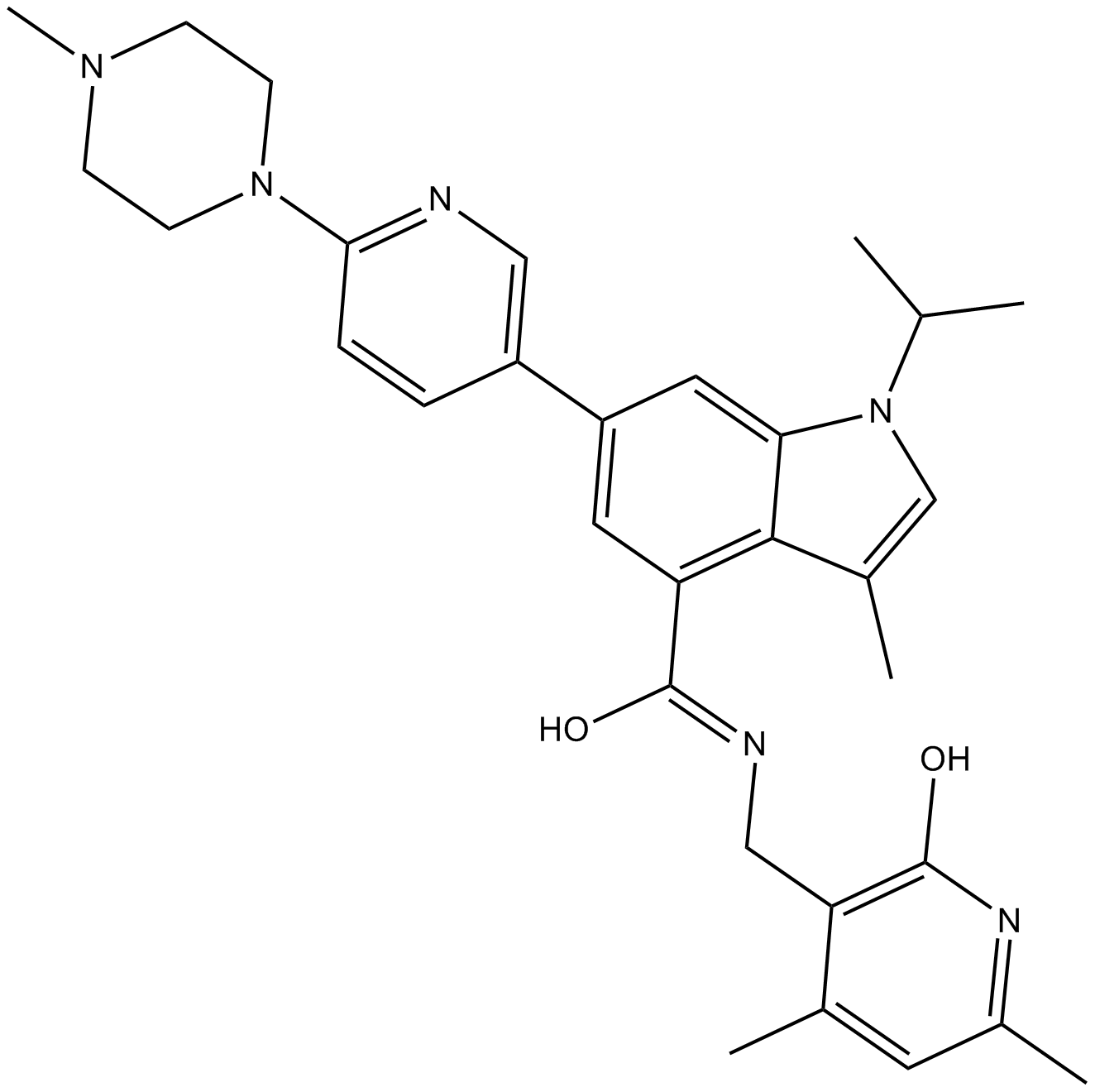
-
GC14585
GSK591
GSK591 (EPZ015866) ist ein potenter und selektiver Inhibitor der Proteinmethyltransferase 5 (PRMT5) mit einem IC50 von 4 nM.
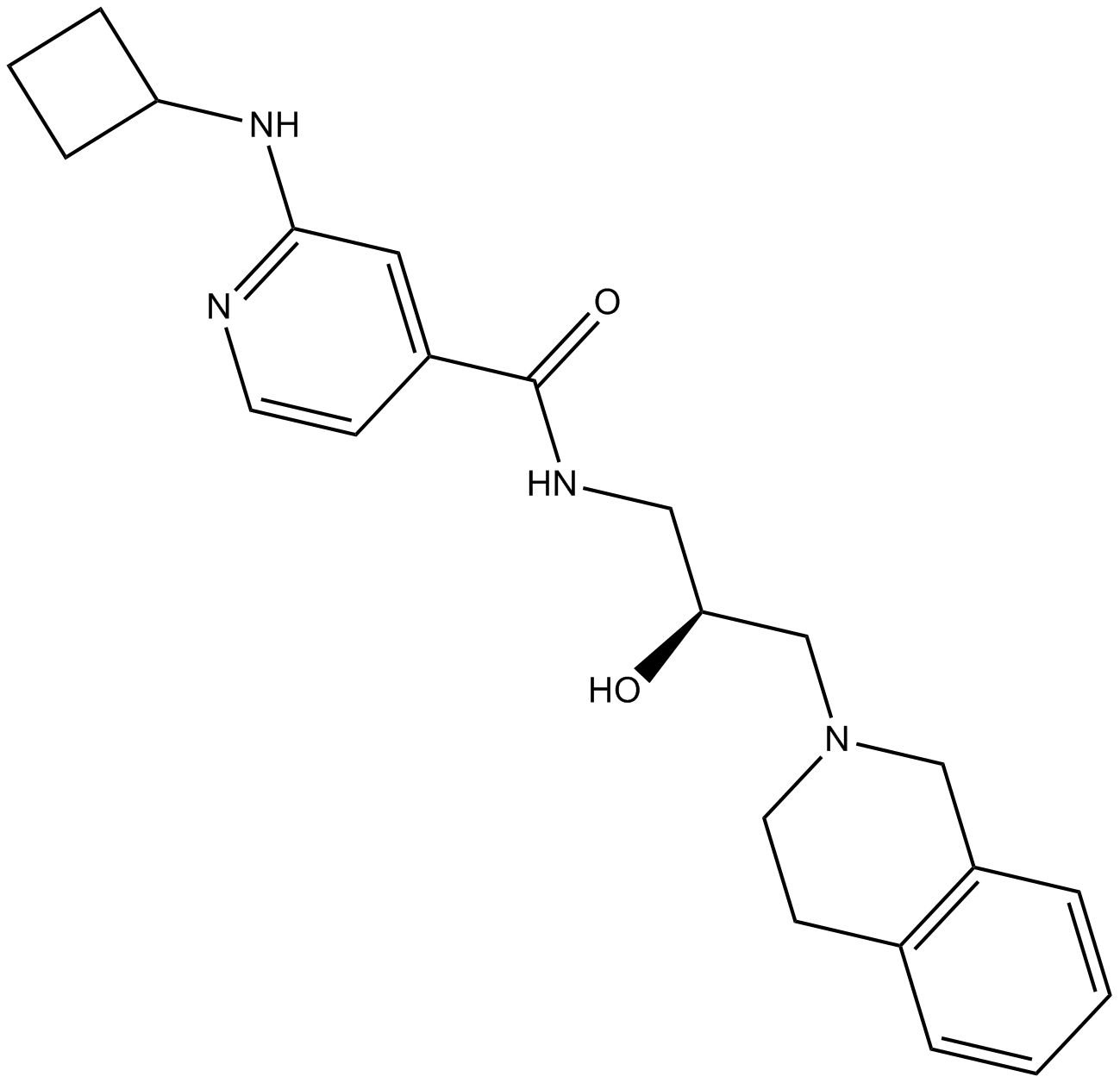
-
GC62312
GSK620
GSK620 ist ein potenter und oral wirksamer Pan-BD2-Inhibitor mit exzellenter breiter Selektivität, Entwickelbarkeit und in vivo oralen Pharmakokinetik.
-
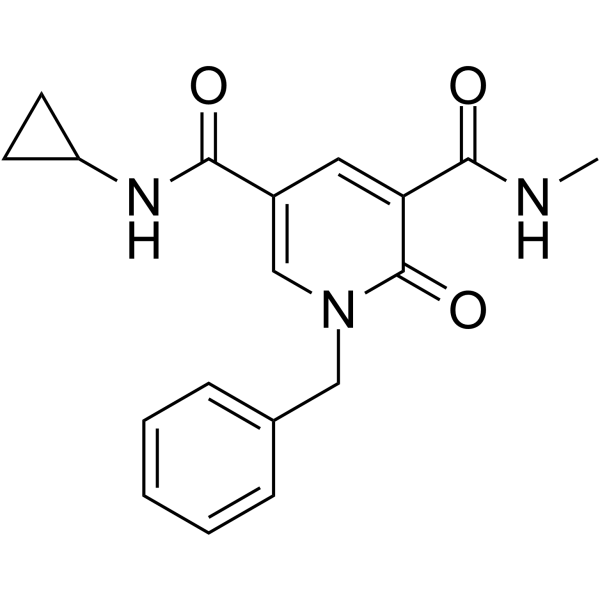
GC13025
GSK6853
GSK6853 ist ein potenter und selektiver Inhibitor der BRPF1-BromodomÄne.
-
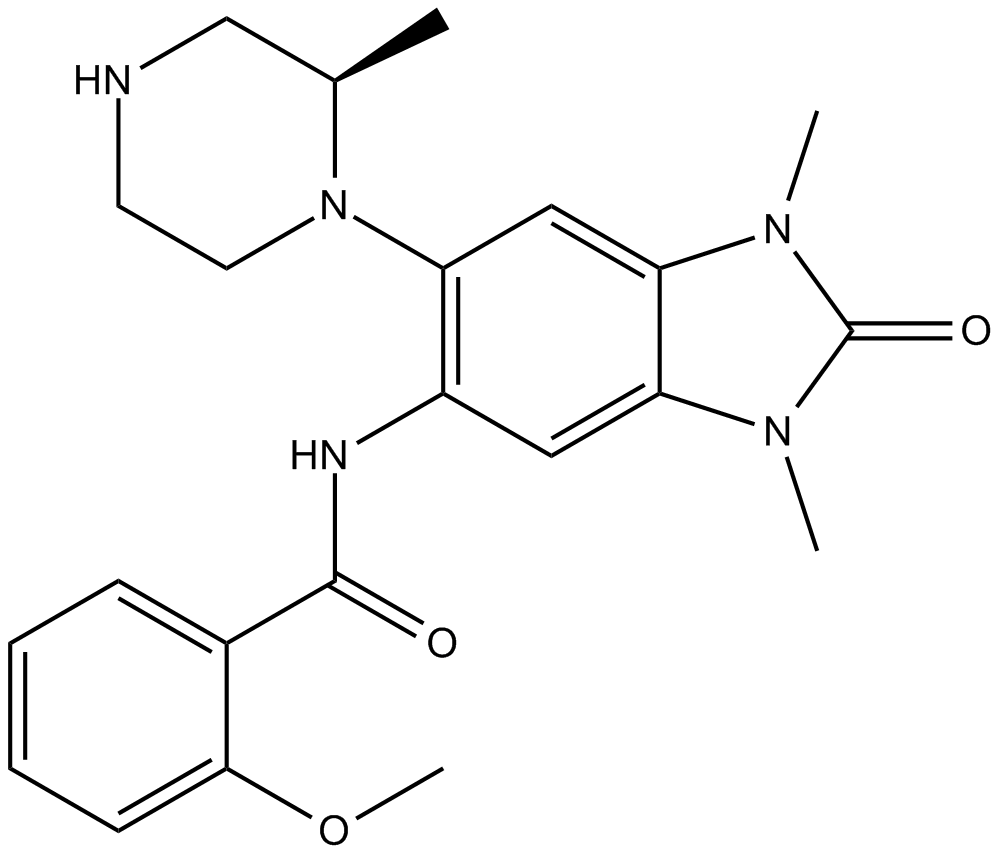
GC62654
GSK778
GSK778 (iBET-BD1) ist ein potenter und selektiver BD1-BromodomÄnen-Inhibitor der BET-Proteine mit IC50-Werten von 75 nM (BRD2 BD1), 41 nM (BRD3 BD1), 41 nM (BRD4 BD1) und 143 nM (BRDT BD1) , beziehungsweise.
-
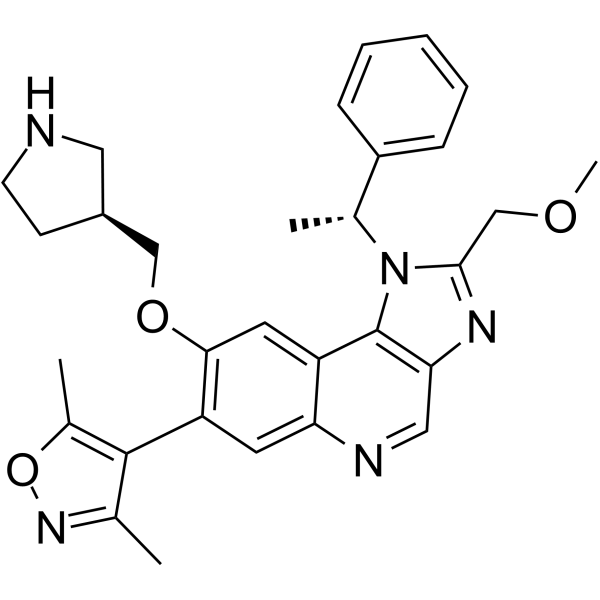
GC38049
GSK8573
GSK8573 ist eine inaktive Kontrollverbindung fÜr GSK2801 (kompetitiver Acetyl-Lysin-Inhibitor der BAZ2A- und BAZ2B-BromdomÄnen). GSK8573 hat BindungsaktivitÄt an BRD9 mit einem Kd-Wert von 1,04 μM und ist inaktiv gegen BAZ2A/B und andere BromodomÄnenfamilien. GSK8573 kann als strukturell verwandte negative Kontrollverbindung in biologischen Experimenten verwendet werden.
-
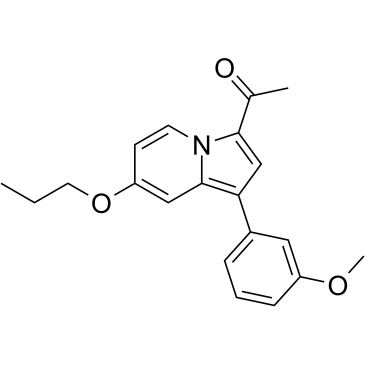
GC60184
GSK8814
GSK8814 ist eine potente, selektive und chemische ATAD2/2B-BromdomÄnensonde und ein Inhibitor mit einer Bindungskonstante von pKd=8,1 und einem pKi=8,9 in BROMOscan. GSK8814 bindet an ATAD2 und BRD4 BD1 mit pIC50s von 7,3 bzw. 4,6. GSK8814 zeigt eine 500-fache SelektivitÄt fÜr ATAD2 gegenÜber BRD4 BD1.
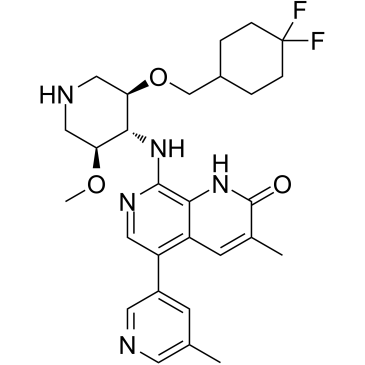
-
GC33324
GSK9311
GSK9311, ein weniger aktives Analogon von GSK6853, kann als negative Kontrolle verwendet werden. GSK9311 hemmt die BRPF-BromodomÄne mit pIC50-Werten von 6,0 und 4,3 fÜr BRPF1 bzw. BRPF2.
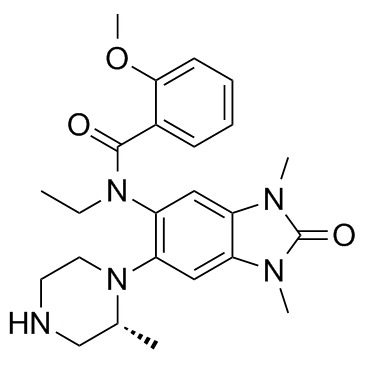
-
GC36196
Guadecitabine sodium
Guadecitabin ist ein neuartiges hypomethylierendes Dinukleotid aus Decitabin und Desoxyguanosin, das gegen den Abbau durch Cytidindeaminase resistent ist.
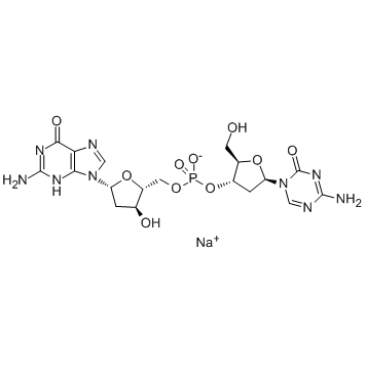
-
GC33041
Gusacitinib (ASN-002)
Gusacitinib (ASN-002) (ASN-002) ist ein oral aktiver und potenter dualer Inhibitor der Milz-Tyrosin-Kinase (SYK) und der Janus-Kinase (JAK) mit IC50-Werten von 5-46 nM. Gusacitinib (ASN-002) wirkt sowohl bei soliden als auch bei hÄmatologischen Tumorarten gegen Krebs.
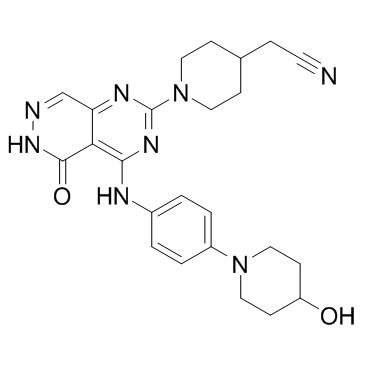
-
GC16611
GW841819X
BET bromodomain inhibitor
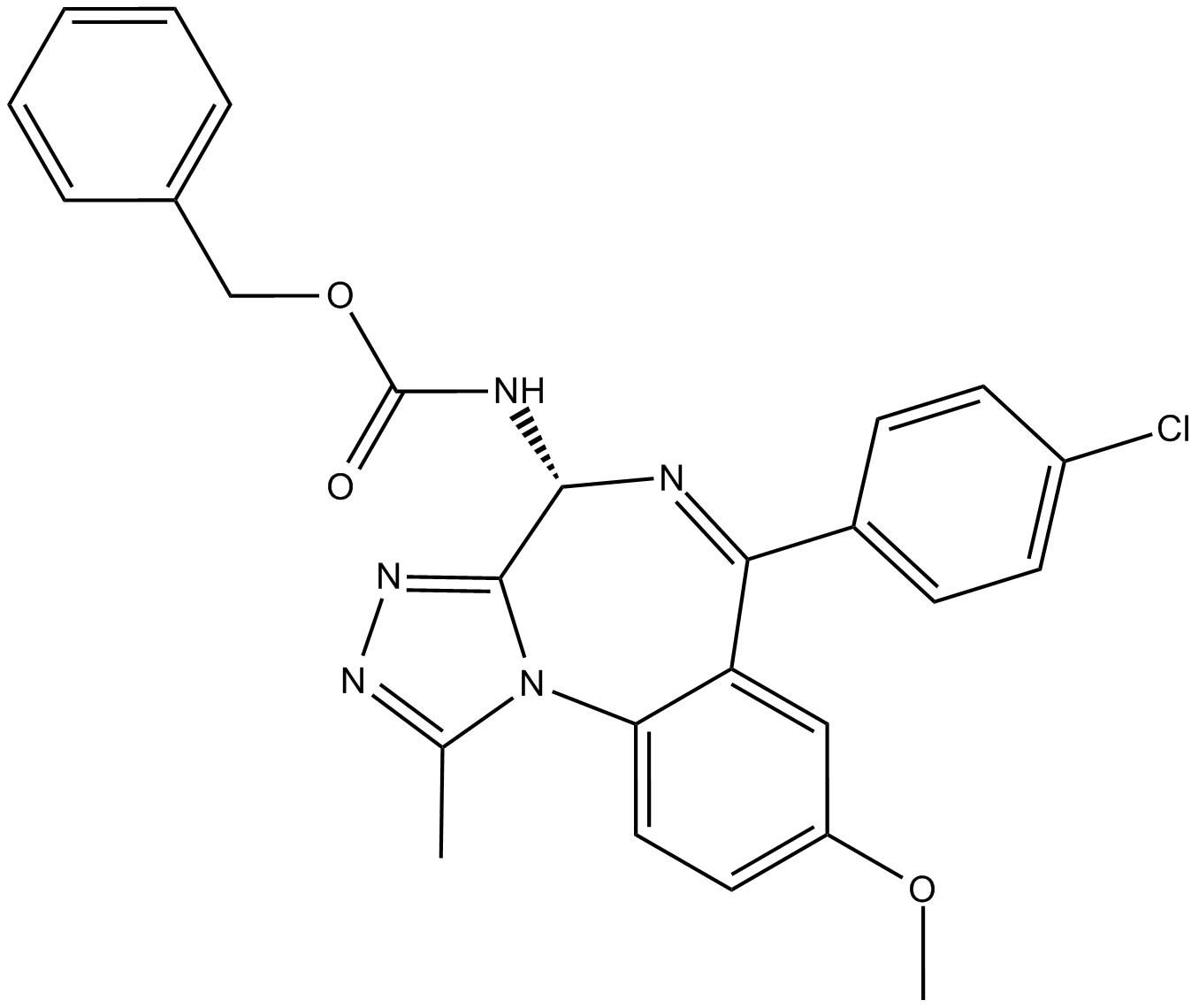
-
GC64115
Gypenoside LI
Gypenosid LI, ein Gypenosid-Monomer, besitzt AntitumoraktivitÄt. Gypenosid LI induziert Zellapoptose, Zellzyklus und Migration.
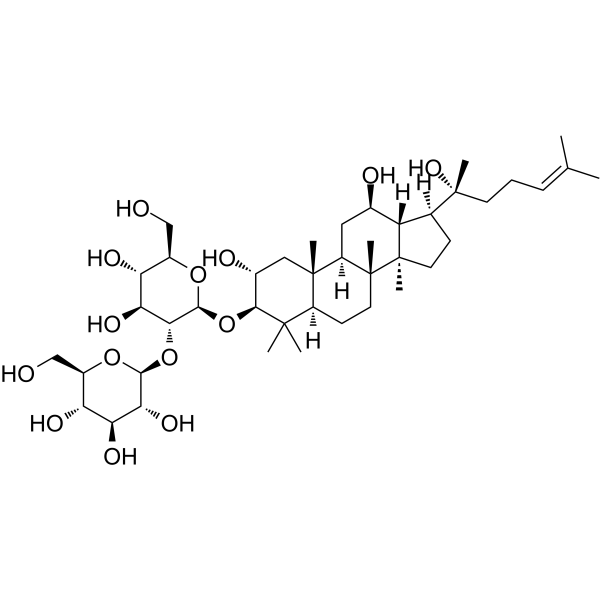
-
GC12009
HAT Inhibitor II
HAT-Inhibitor II (Verbindung 2c) ist ein potenter, selektiver und zellgÄngiger p300-Histon-Acetyltransferase-Inhibitor mit einem IC50-Wert von 5 μM. HAT-Inhibitor II zeigt Anti-Acetylase-AktivitÄt in SÄugerzellen.
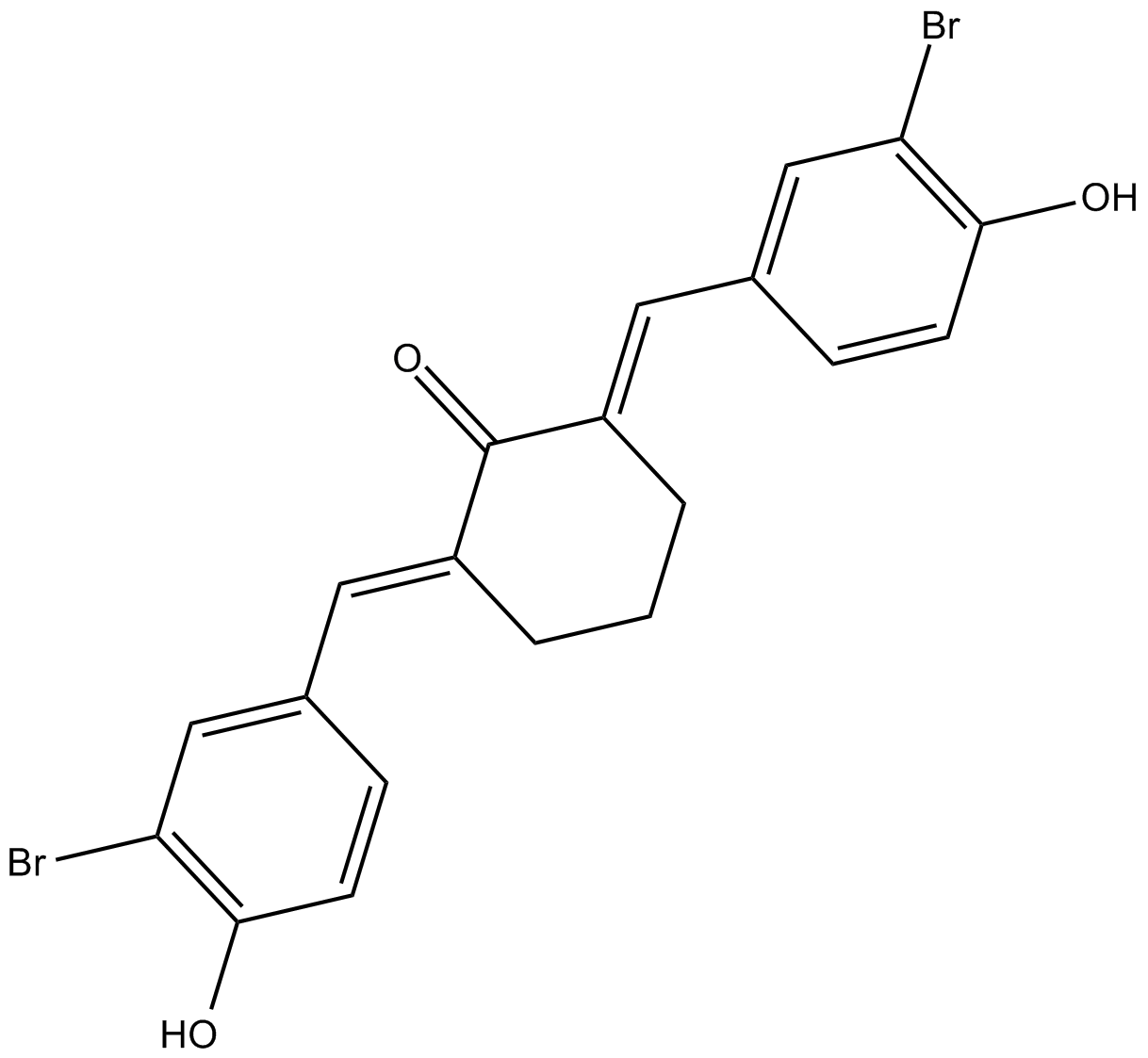
-
GC33422
HAT-IN-1
HAT-IN-1 ist ein Inhibitor von HAT, der in der Krebsforschung eingesetzt wird.
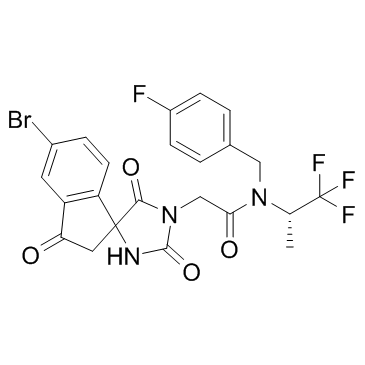
-
GC43806
HC Toxin
HC Toxin, ein zyklisches Tetrapeptid, ist ein potenter HDAC-Inhibitor mit einem IC50 von 30 nM. HC-Toxin induziert die Apoptose von Tumorzellen und hat Antikrebswirkungen.
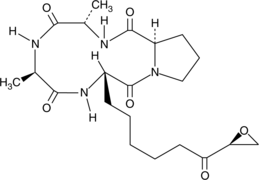
-
GC19190
HDAC-IN-4
HDAC-IN-4 ist ein potenter, selektiver und oral aktiver HDAC-Inhibitor der Klasse I mit IC50-Werten von 63 nM, 570 nM und 550 nM fÜr HDAC1, HDAC2 bzw. HDAC3. HDAC-IN-4 hat keine AktivitÄt gegen HDAC Klasse II. HDAC-IN-4 hat AntitumoraktivitÄt.
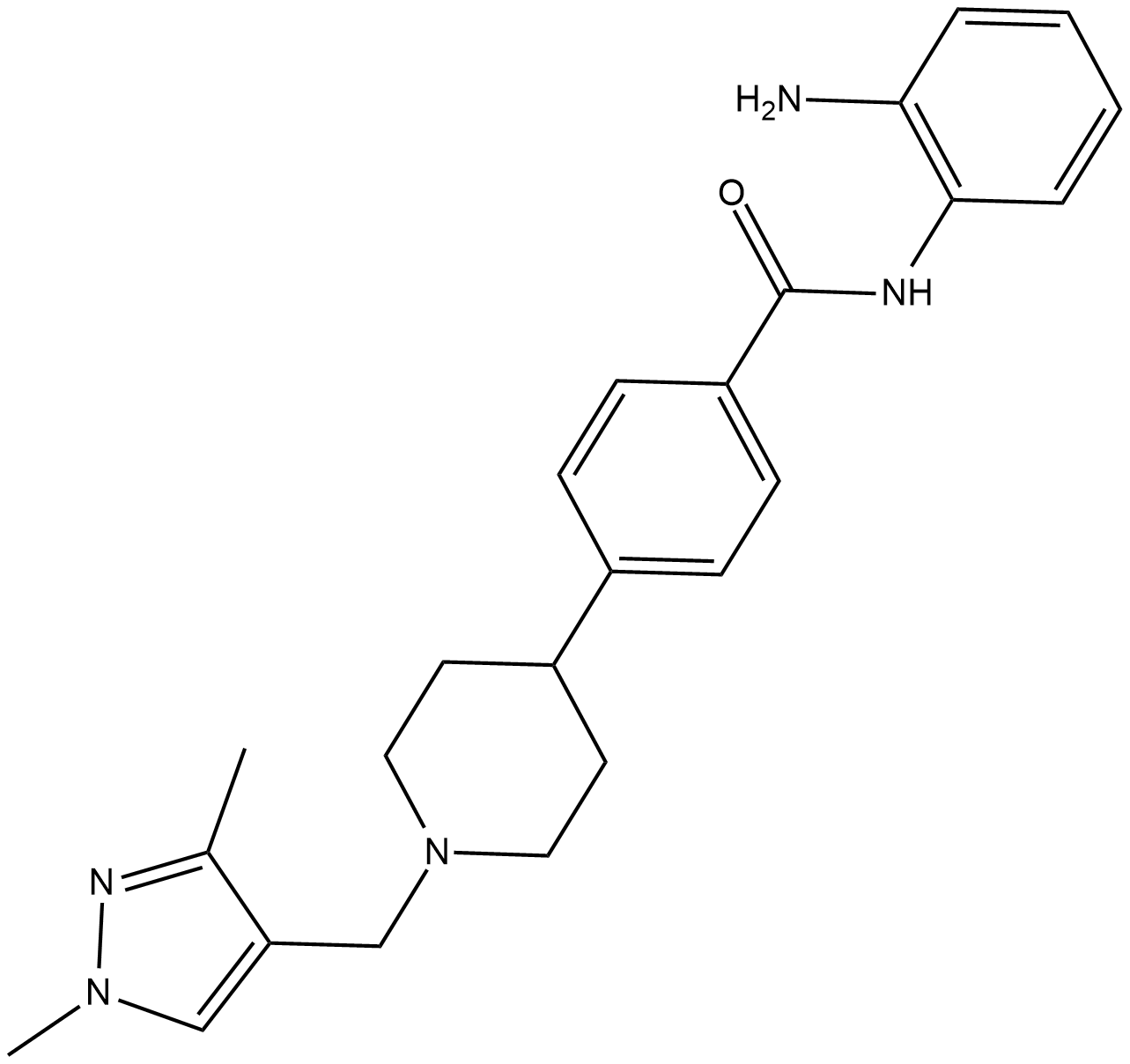
-
GC66052
HDAC-IN-40
HDAC-IN-40 ist ein potenter HDAC-Inhibitor auf Alkoxyamidbasis mit Ki-Werten von 60 nM und 30 nM fÜr HDAC2 bzw. HDAC6. HDAC-IN-40 hatte Antitumorwirkungen.
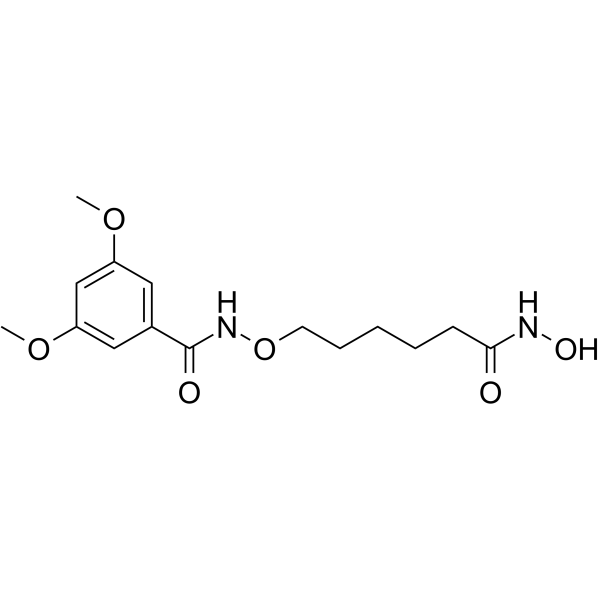
-
GC33395
HDAC-IN-5
HDAC-IN-5 ist ein Inhibitor der Histon-Deacetylase (HDAC).
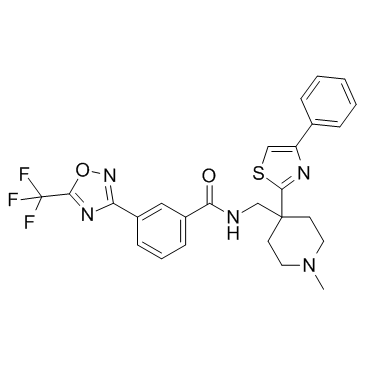
-
GC41495
HDAC3 Inhibitor
HDAC3-Inhibitor (Verbindung 5) ist ein potenter und selektiver HDAC3-Inhibitor mit einem IC50-Wert von 5,96 nM.
-
GC68010
HDAC3-IN-T247
-
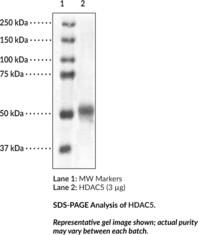
GC49693
HDAC5 (human, recombinant)
Active, pure human recombinant enzyme
-
GC41085
HDAC6 Inhibitor
HDAC6-Inhibitor ist ein potenter und selektiver HDAC6-Inhibitor (IC50=36 nM). Der HDAC6-Inhibitor hemmt andere HDAC-Isoformen schwach. HDAC6-Inhibitor hemmt die Acyl-Tubulin-Akkumulation in Zellen mit einem IC50-Wert von 210 nM.
-
GC33317
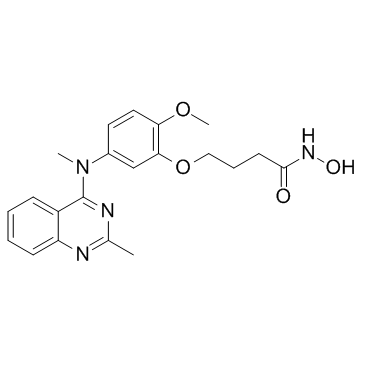
HDAC6-IN-1
HDAC6-IN-1 ist ein potenter und selektiver Inhibitor fÜr HDAC6 mit einem IC50 von 17 nM und zeigt eine 25-fache bzw. 200-fache SelektivitÄt im Vergleich zu HDAC1 (IC50=422 nM) bzw. HDAC8 (IC50=3398 nM).
-
GC19189
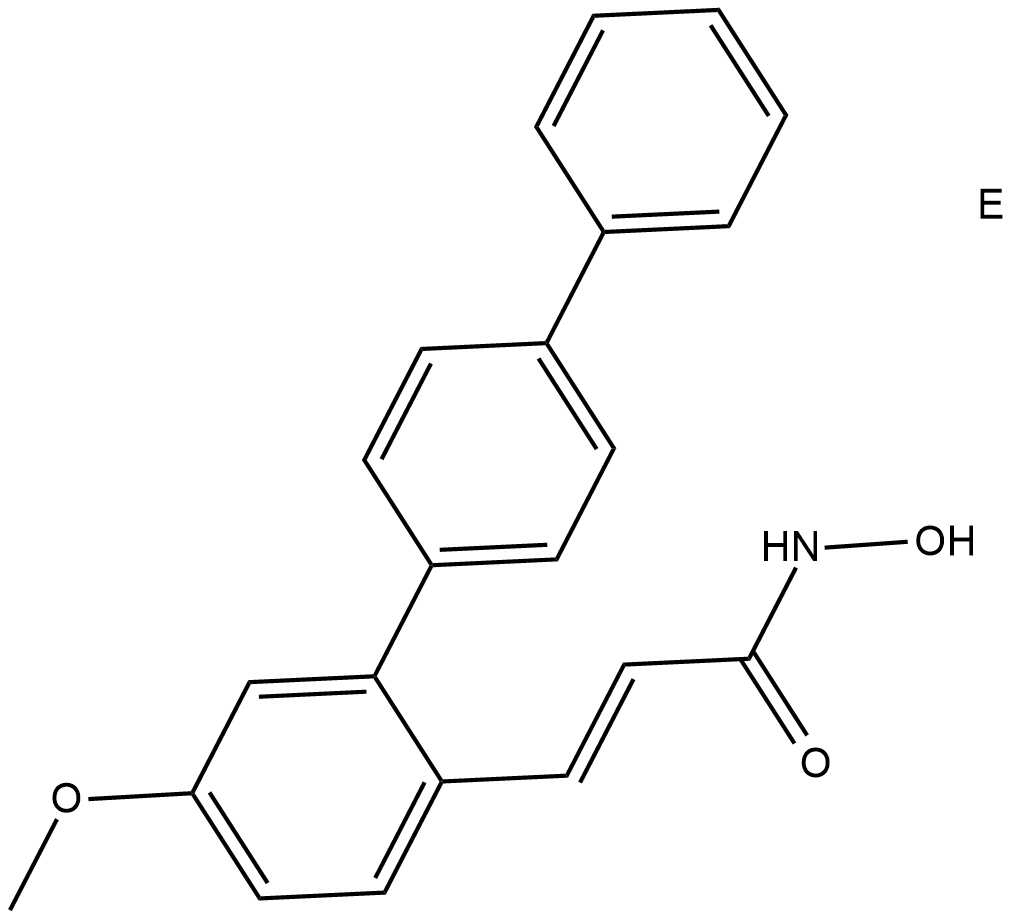
HDAC8-IN-1
HDAC8-IN-1 ist ein HDAC8-Inhibitor mit einem IC50 von 27,2 nM.
-
GC65460
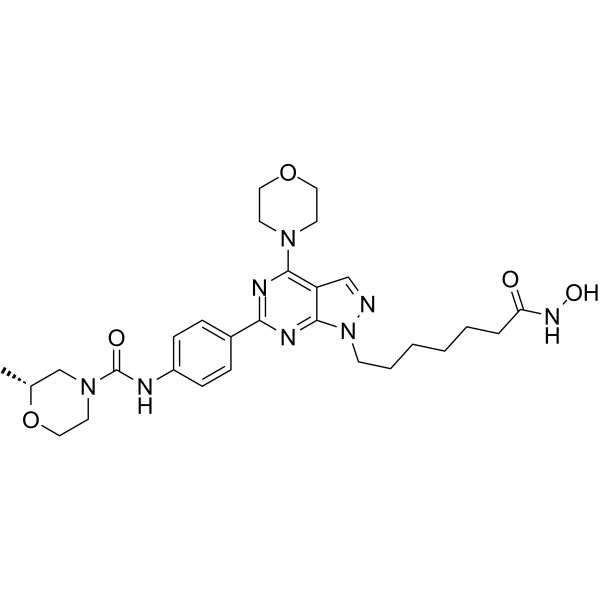
HDACs/mTOR Inhibitor 1
HDACs/mTOR Inhibitor 1 ist ein dualer Histon-Deacetylasen (HDACs)- und SÄugetier-Target-Inhibitor von Rapamycin (mTOR) zur Behandlung hÄmatologischer Malignome mit IC50-Werten von 0,19 nM, 1,8 nM, 1,2 nM und >500 nM fÜr HDAC1, HDAC6, mTOR und PI3Kα. HDACs/mTOR Inhibitor 1 stimuliert den Zellzyklusarrest in der G0/G1-Phase und induziert die Apoptose von Tumorzellen mit geringer ToxizitÄt in vivo.
-
GC17196
Hesperadin
A multi-kinase inhibitor
-
GC50050
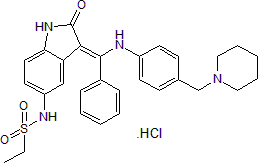
Hesperadin hydrochloride
Hesperadin-Hydrochlorid ist ein ATP-kompetitiver Indolinon-Inhibitor von Aurora A und B. Hesperadin-Hydrochlorid hemmt Aurora B mit einem IC50 von 250 nM.
-
GC39146
HIF-1 inhibitor-1
An inhibitor of HIF-1 signaling

-
GC66464
HIF-1α-IN-2
HIF-1α-IN-2 ist ein wirksames HIF-1&7#945; Inhibitor mit Antikrebspotenzen (IC50s von 28 nM und 15 nM in MDA-MB-231- bzw. MiaPaCa-2-Zellen). HIF-1α-IN-2 unterdrÜckt HIF-1α Expression durch Blockieren der Transkription und Proteintranslation.
-
GC62584
HIF-2α-IN-2
HIF-&2#945;-IN-2 ist ein Hypoxie-induzierbarer Faktor (HIF-2α)-Inhibitor, extrahiert aus dem Patent WO2015035223A1, Verbindung 232, hat einen IC50 von 16 nM im Scintillation Proximity Assay (SPA).
-
GC62418
HIF-2α-IN-3
HIF-2α-IN-3, ein allosterischer Inhibitor des Hypoxie-induzierbaren Faktors-2α (HIF-2α), weist einen IC50 von 0,4 μM und einen KD von 1,1 μM auf. Antikrebsmittel.
-
GC63678
HIF-2α-IN-4
HIF-2α -IN-4 ist ein potenter Inhibitor des Hypoxie-induzierbaren Faktors-2α (HIF-2α) Übersetzung, mit einem IC50 von 5 μM. HIF-2α -IN-4 verringert sowohl konstitutives als auch Hypoxie-induziertes HIF-2α Proteinexpression. HIF-2α-IN-4 verknÜpft sein auf Eisen ansprechendes Element 5'UTR mit der Sauerstoffmessung.
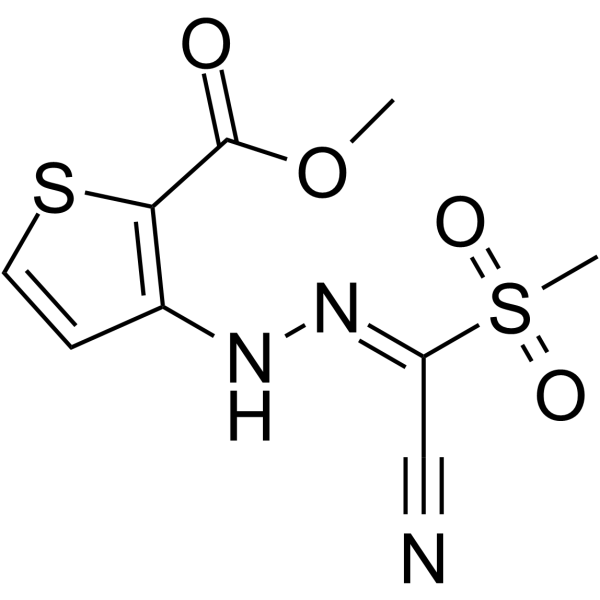
-
GC31358
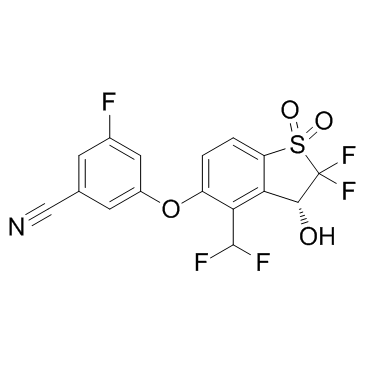
HIF-2α-IN-1
HIF-&2alpha;-IN-1 ist ein HIF-2α-Inhibitor mit einem IC50-Wert von weniger als 500 nM im HIF-2α-Szintillations-Proximity-Assay.
-
GC67941
HIF-PHD-IN-1
-
GC65570
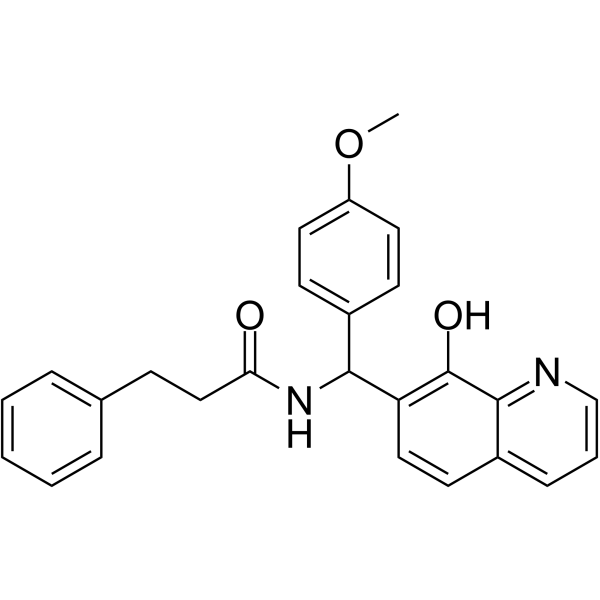
HIF1-IN-3
HIF1-IN-3 (Verbindung F4) ist ein potenter HIF1-Inhibitor mit einem EC50-Wert von 0,9 μM. HIF1-IN-3 kann fÜr die Erforschung von Krebserkrankungen verwendet werden.
-
GC11302
Hinokitiol
Hinokitiol ist ein Bestandteil Ätherischer Öle, die aus Chymacyparis obtusa isoliert wurden, reduziert die Nrf2-Expression und verringert die mRNA- und Proteinexpression von DNMT1 und UHRF1 mit antiinfektiÖsen, antioxidativen und AntitumoraktivitÄten.

-
GC12359
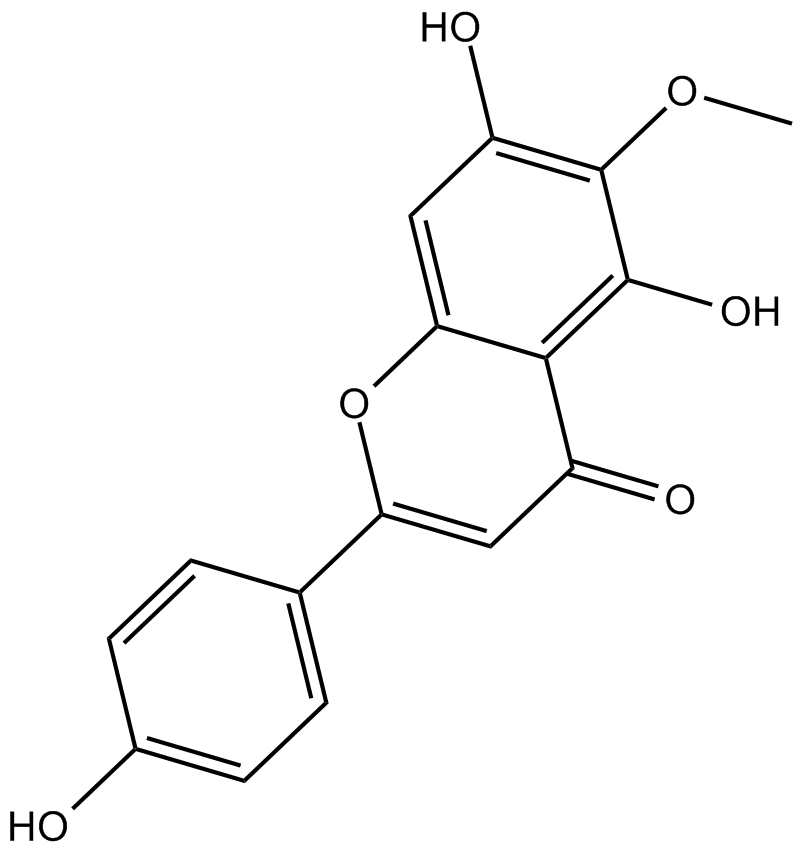
Hispidulin
Hispidulin ist ein natÜrliches Flavon mit einem breiten Spektrum an biologischen AktivitÄten. Hispidulin ist ein Pim-1-Inhibitor mit einem IC50 von 2,71 μM.
-
GC43831
Histone H3 (21-44)-GK-biotin (trifluoroacetate salt)
Histone H3 (21-44)-GK-biotin is a peptide fragment of histone H3 that corresponds to amino acid residues 22-45 of the human histone H3.1 and 3.2 sequences and is biotinylated via a C-terminal GK linker.

-
GC43832
Histone H3 (21-44)-GK-biotin amide (trifluoroacetate salt)
Histone H3 (21-44)-GK-biotin is a peptide fragment of histone H3 that corresponds to amino acid residues 22-45 of the human histone H3.3 sequence and is biotinylated via a C-terminal GK linker.
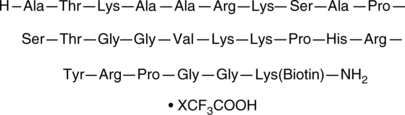
-
GC52479
Histone H3 (Citrullinated R26) (21-44)-GGK-biotin Peptide (trifluoroacetate salt)
A biotinylated peptide fragment of histone H3
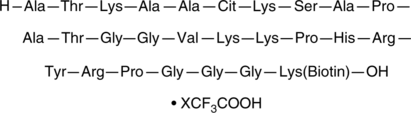
-
GC43846
Histone H3K27Me1 (23-34) (trifluoroacetate salt)
Histone H3K27Me1 (23-34) is a peptide fragment of histone H3 that corresponds to amino acid residues 24-35 of the human histone H3.1 and H3.2 sequences.
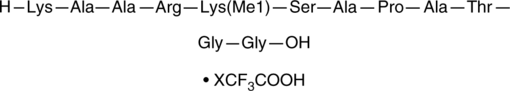
-
GP10020
Histone-H2A-(107-122)-Ac-OH
Histone-H2A peptide
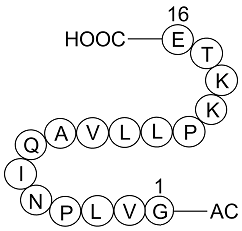
-
GC33211
HJB97
HJB97 ist ein hochaffiner BET-Inhibitor mit Kis von 0,9 nM (BRD2 BD1), 0,27 nM (BRD2 BD2), 0,18 nM (BRD3 BD1), 0,21 nM (BRD3 BD2), 0,5 nM (BRD4 BD1), 1,0 nM (BRD4 BD2), bzw. HJB97 wird fÜr das Design eines potenziellen PROTAC BET-Abbaumittels verwendet und hat AntitumoraktivitÄt.
-
GC17023
HLCL-61
HLCL-61 ist ein erstklassiger Inhibitor der Protein-Arginin-Methyltransferase 5 (PRMT5).
-
GC12334
HNHA
HNHA ist ein potenter Inhibitor der Histon-Deacetylase (HDAC). HNHA hÄlt den Zellzyklus in der G1/S-Phase Über p21-Induktion an. HNHA hemmt das Tumorwachstum und die Tumorneovaskularisation. HNHA kann ein wirksames Antikrebsmittel gegen Brustkrebs sein.
-
GC11574
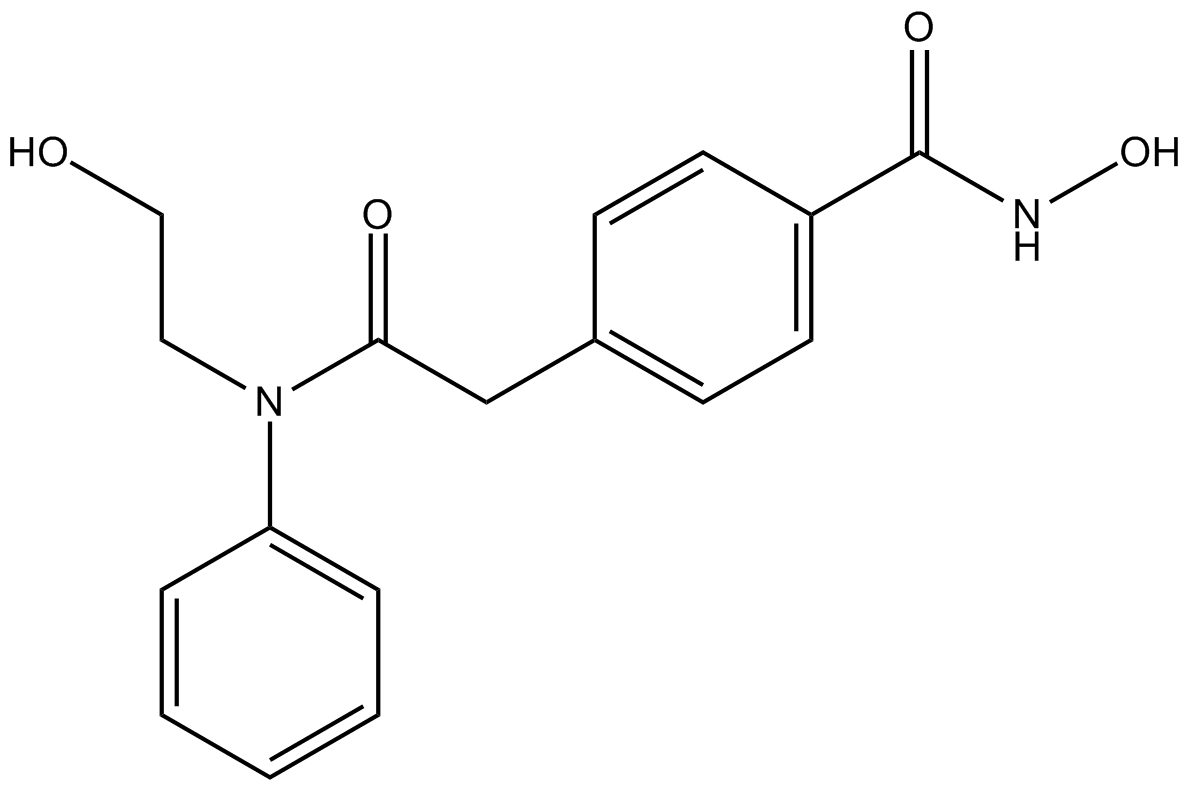
HPOB
HPOB ist ein hochpotenter und selektiver Inhibitor von HDAC6 mit einem IC50 von 56 nM. HPOB zeigt eine >30-fach geringere Wirksamkeit gegenÜber anderen HDACs. HPOB verstÄrkt die Wirksamkeit von DNA-schÄdigenden Antikrebsmitteln in transformierten Zellen, aber nicht in normalen Zellen. HPOB blockiert nicht die Ubiquitin-bindende AktivitÄt von HDAC6.
-
GC11767
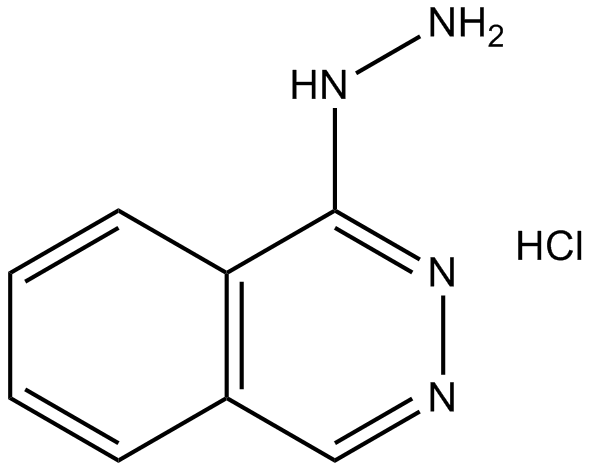
Hydralazine HCl
Hydralazin HCl ist ein oral wirksames Antihypertensivum, das den peripheren Widerstand direkt durch Entspannung der glatten Muskelzellschicht in arteriellen GefÄßen reduziert.
-
GC60919
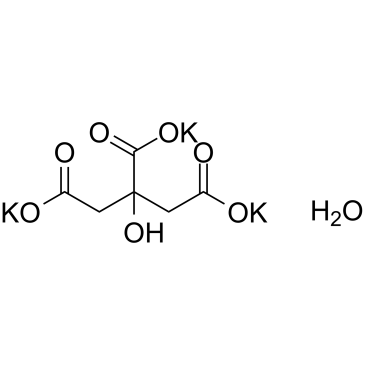
Hydroxycitric acid tripotassium hydrate
HydroxyzitronensÄure-Trikaliumhydrat (Kaliumcitrat-Monohydrat) ist der Hauptwirkstoff von Garcinia Cambogia.
-
GC50070
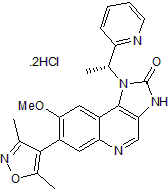
I-BET 151 dihydrochloride
I-BET 151-Dihydrochlorid (GSK1210151A-Dihydrochlorid) ist ein BET-Bromodomänen-Inhibitor, der BRD4, BRD2 und BRD3 mit pIC50 von 6,1, 6,3 bzw. 6,6 hemmt.
-
GC13187
I-BET 151 hydrochloride
BET bromodomain inhibitor
-
GC17073
I-BET-762
I-BET-762 (I-BET762; GSK525762) ist ein BET-BromodomÄnen-Inhibitor mit IC50 von 32,5-42,5 nM.
-
GC15747
I-BET151 (GSK1210151A)
I-BET151 (GSK1210151A) (GSK1210151A) ist ein BET-BromodomÄnen-Inhibitor, der BRD4, BRD2 und BRD3 mit pIC50 von 6,1, 6,3 bzw. 6,6 hemmt.
-
GC64297
I-BET567
I-BET567 ist ein potenter und oral aktiver Inhibitor des Pan-BET-Kandidaten mit pIC50-Werten von 6,9 und 7,2 fÜr BRD4 BD1 bzw. BD2.
-
GC12588
I-BRD9
I-BRD9 ist eine selektive zellulÄre chemische Sonde des BromodomÄnen-enthaltenden Proteins 9 (BRD9) mit einem pIC50-Wert von 7,3 μM.
-
GC17944
I-CBP 112
A p300 and CBP inhibitor
-
GC34078
I-CBP112
I-CBP112 ist ein spezifischer und potenter Acetyl-Lysin-kompetitiver Protein-Protein-Interaktionsinhibitor, der die CBP/p300-BromodomÄnen hemmt und die Acetylierung durch p300 verstÄrkt.